Nature | 燕麦泛基因组再登顶主刊
燕麦Avena sativa是全球第七大谷物,因其高纤维含量对人类健康有益。尽管燕麦具有提高产量和可持续性种植的潜力,但由于其复杂的基因组结构,遗传改良进展较慢。燕麦是一种同种异体六倍体,包含A、C、D三个亚基因组,每个亚基因组大小在3Gb到4Gb之间。与许多其他谷物物种相比,燕麦的基因组研究仍处于早期阶段,对结构基因组多样性和基因表达变异性的研究仍然不足。
加拿大研究团队于2025年10月29日在国际著名期刊Nature上发表了一篇题目为“A pangenome and pantranscriptome of hexaploid oat”的研究论文,该研究通过组装33个野生和驯化燕麦品种的基因组并进行转录组联合分析,系统地鉴定了燕麦各品种中的结构变异及其对表型的影响,为未来燕麦的遗传改良和育种工作奠定了重要的基础。

一、六倍体燕麦的全基因组注释
研究团队组装并注释了33个不同燕麦品系的基因组,涵盖了来自主要燕麦种植区的优良品种、野生燕麦样本及其亲缘物种,如A. longiglumis、A. insularis,以及合成六倍体Amagalon等。基因组的组装和注释采用了PacBio HiFi平台和Illumina短读测序技术,平均99.97%的序列被精确地分配到染色体上。通过RNA-seq和PacBio Iso-Seq对23个品系的转录组进行测序,并结合蛋白质同源性和ab initio预测,完成了基因的注释。针对六倍体燕麦,研究预测了107847到136836个基因,其中约60.5%为表达基因。通过BUSCO评估基因注释的完整性,结果显示98.8%的基因为完整,且短读组装的完整性优于长读组装。
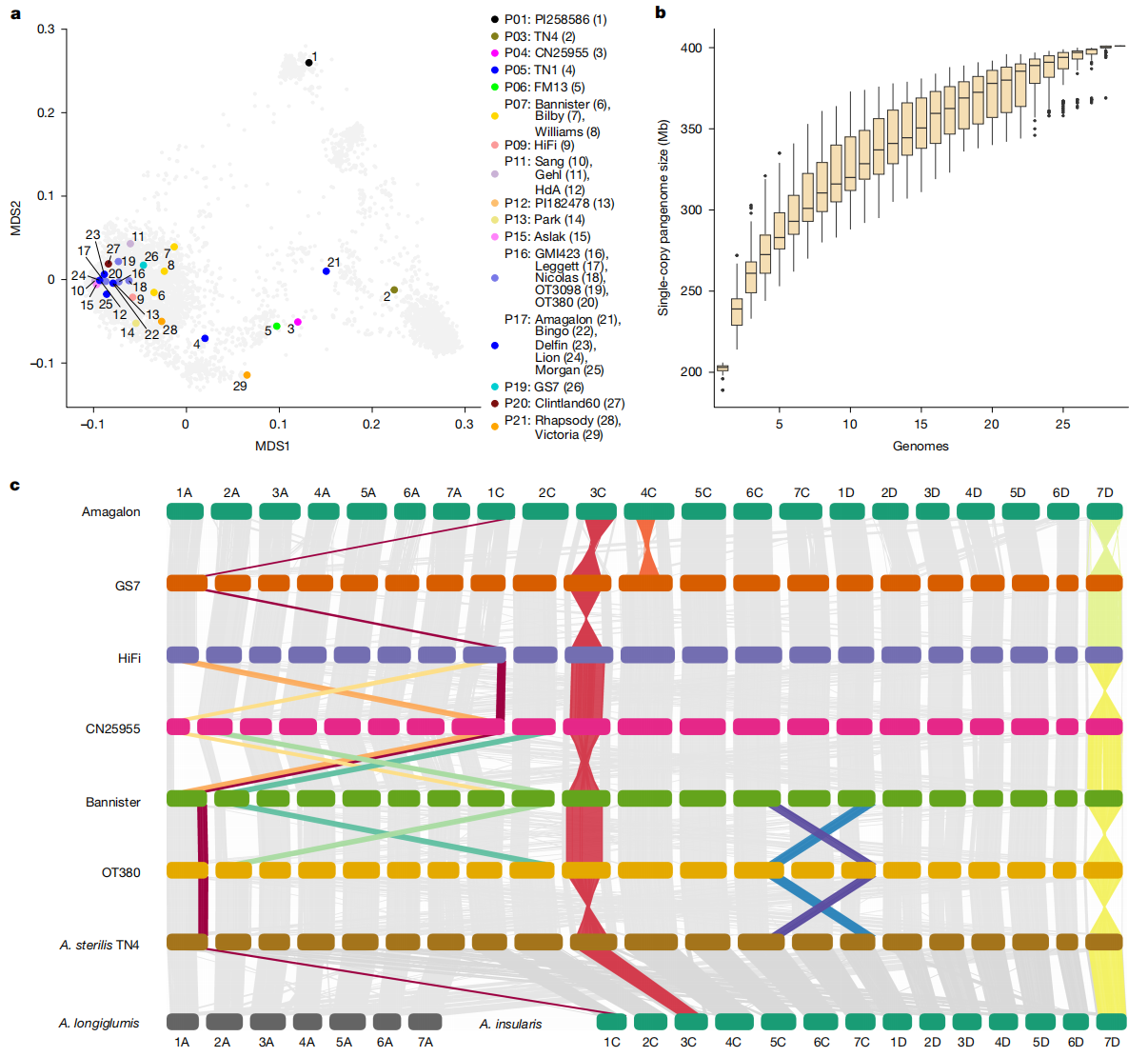
图1:燕麦泛基因组组成和结构变异
二、基于基因的PAV目录
作者通过Orthofinder构建基因同源框架,分析了泛基因组中的基因多态性(PAV)。泛基因组的基因可分为核心基因、壳基因和云基因:核心基因在所有基因组中均有存在,壳基因在多个基因组中存在,云基因则仅在一个基因组中存在。统计结果显示,核心基因组富含与植物基本生理过程相关的基因,如花形成和营养物质吸收,而壳基因组则与防御机制和种子储存有关。云基因则主要涉及植物防御和离子转运等关键过程。进一步分析发现,核心基因在多倍体燕麦基因组中的表达水平高于云基因和壳基因,且在C基因组中的基因表达量显著低于A和D基因组。

图2:燕麦泛转录组
三、基因表达动力学的多样性
本研究利用23个燕麦品系的RNA测序数据,系统揭示了燕麦泛转录组在亚基因组、组织及品系间的表达动态多样性。研究聚焦于20个六倍体燕麦中在A、C、D三个亚基因组均为单拷贝的5965个基因,发现约49.4%的基因在不同品系中表现出一致的表达模式,被定义为“稳定三联体”。这些基因在A、C、D三个亚基因组间的表达相对平衡,主要参与蛋白质合成、囊泡运输等基本细胞功能;少部分基因表现出表达偏好,主要集中在C亚基因组。从组织层面看,在叶片、节间和颖果中的表达差异较大,而根、胚和花序的表达相对保守。进一步分析表明,基因表达多样性与DNA序列变异(如启动子或调控元件差异)密切相关。仅13.5%的基因在所有组织和品系中保持稳定,显示出明显的组织特异性调控。部分亚基因组基因缺失时,A与D亚基因组的同源基因常发生代偿性上调,而C亚基因组代偿能力较弱,反映出A与D亚基因组亲缘更近、功能互补性更强。
四、结构变异图谱
六倍体燕麦的镶嵌型基因组源于长期进化过程中积累的罕见染色体重排事件。研究发现,栽培燕麦及其野生祖先A. sterilis中存在多种多态性染色体重排,包括1C→1A与6C→1D的易位及3C染色体的近着丝粒倒位。此外,泛基因组分析还鉴定出四种此前未知的结构变异,如2A/2C易位、4C和7D染色体的大规模近着丝粒倒位等。这些变异多在驯化前形成,并在不同A. sterilis材料中表现出等位差异。结构重排导致断点附近的基因表达发生局部变化,尤其影响代谢与调控相关基因,可能在功能上具有潜在作用。进一步的案例分析表明,7D染色体倒位与开花期调控、2A/2C易位与诱变育种密切相关。这些结构变异不仅存在于野生或古老种质中,也广泛分布于现代栽培品种,持续塑造燕麦基因组结构并影响现代育种进程。
五、与早抽穗相关的染色体倒位
在早期研究中发现7D染色体多数区域存在重组抑制,可能有倒位事件。作者通过对295个燕麦品种的基因组重测序分析,鉴定出两种近着丝粒单倍型位点,可有效区分倒位型与正常型材料。结果显示,携带倒位祖先型单倍型1的材料平均比携带衍生型单倍型2的材料早开花约3.7天,表明该倒位显著影响抽穗期。进一步的QTL定位结果显示,7D染色体上存在与开花时间相关的三个主要基因区域(CO、VRN3/FT 和 Di)。在显著关联区间(468.7-469.8 Mb)中,研究发现关键开花调控基因 FT1/VRN3,其在倒位材料中表达显著上调。结构变异分析还发现,倒位型材料中 FT1-7D 存在18 bp缺失,而 FT1-7A 中的12 bp缺失导致提前终止密码子。综合分析表明,7D染色体倒位可能通过抑制基因间重组或改变开花调控基因的表达模式来调节开花时间,是影响燕麦早熟性的重要遗传结构变异,为改良燕麦生育期性状提供了关键线索。
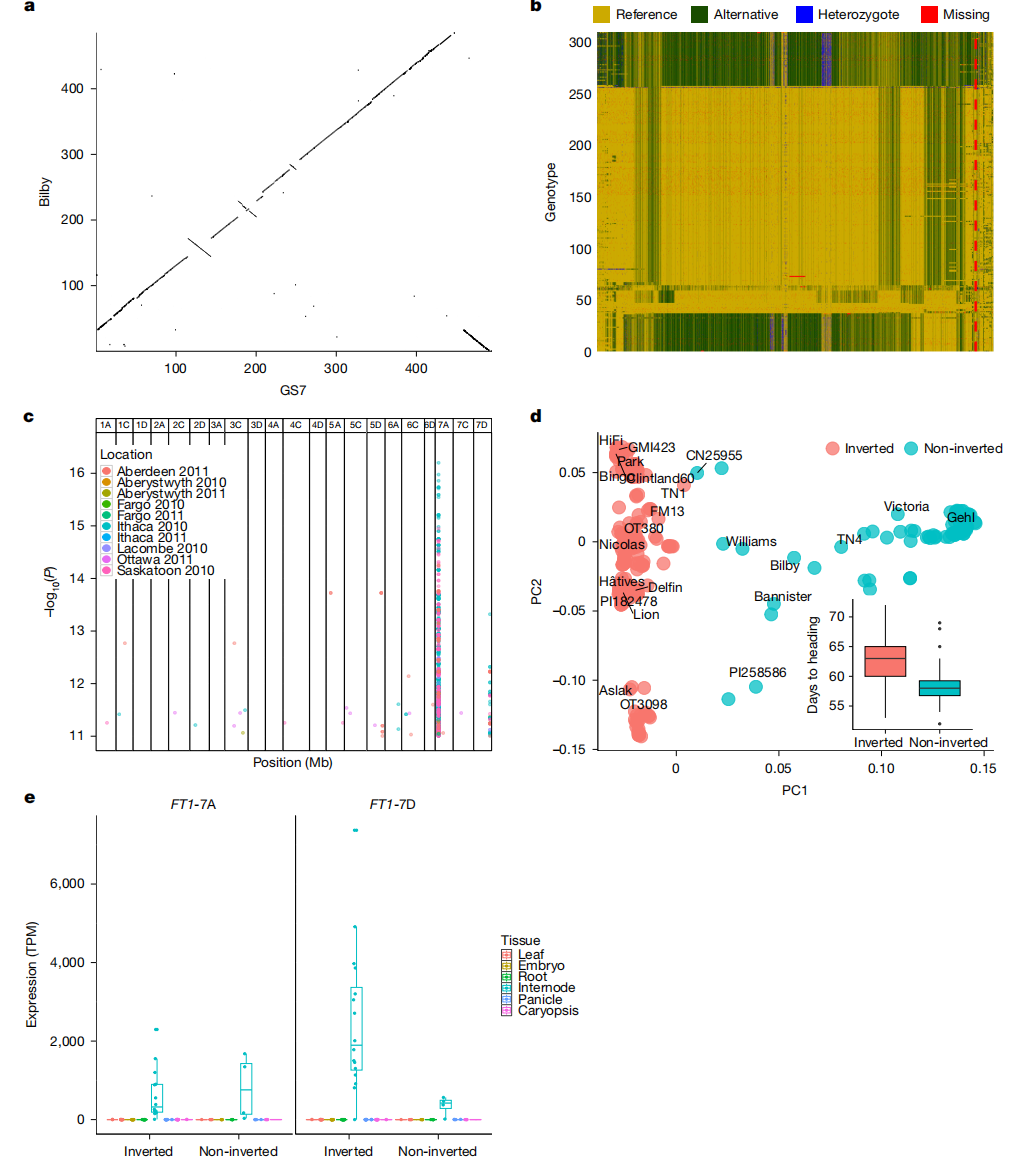
图3 :与提早抽穗有关的染色体倒位
六、突变育种的隐藏遗产
作者进一步对澳大利亚燕麦品种中发现的2A/2C染色体易位进行系统分析,该易位最早出现在1970年通过快中子诱变获得的突变材料OT207中,原本是诱变的偶然副产物,却在之后多个澳大利亚品种中被广泛保留。研究对41个国家564份样本的分型数据分析发现,约3%的材料携带该易位,主要集中于澳大利亚高产品种,如Bannister、Possum和Wombat。而进一步对2017-2022年全国区域试验数据分析显示,携带该易位的品种平均产量显著高于未易位品种,说明该结构可能在澳大利亚环境中具有适应优势。为了验证这一假设及其潜在风险,作者构建了Bannister×Williams的重组系群体,发现部分杂合后代在2A与2C染色体上出现臂缺失与重复,导致生育力下降和籽粒易脱落,说明在杂合状态下该易位可能引发减数分裂异常。此外,作者将株高相关的QTL精确定位到6D染色体上的554kb区间,并发现半矮秆品种共享一个含9个基因的Dw6单倍型,其中可能影响脂肪酸代谢的基因为半矮秆性状的候选基因。
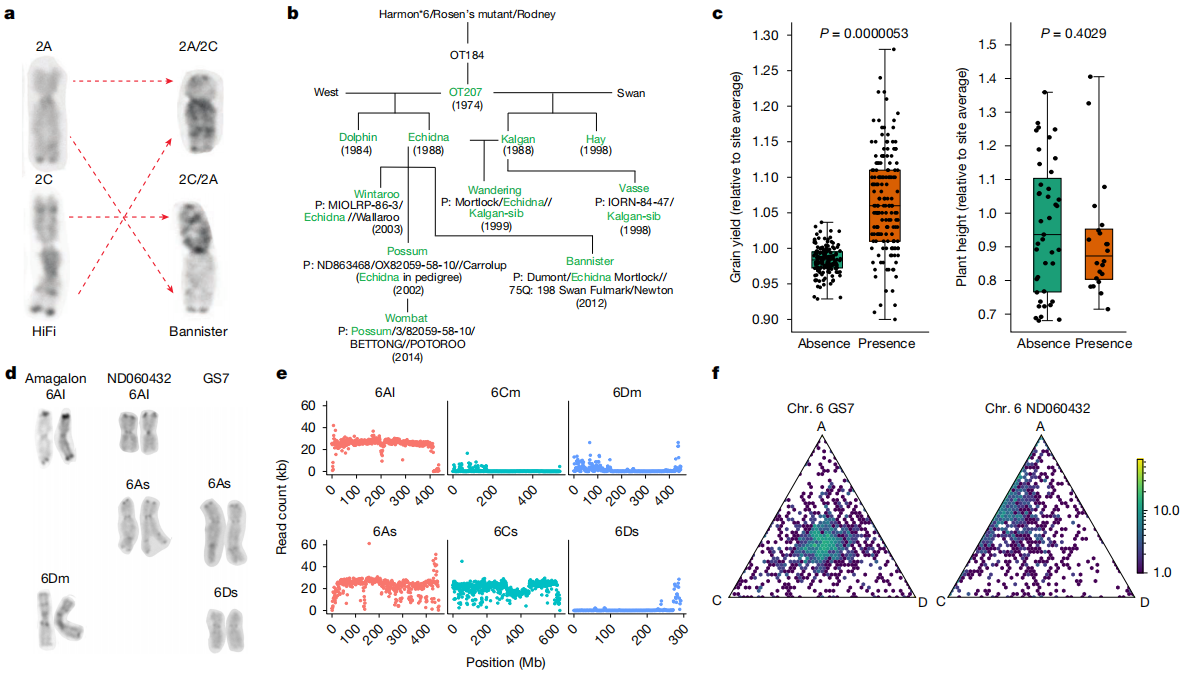
图4:染色体易位和同源序列交换
七、人工合成燕麦的基因组变化
Amagalon由二倍体Avena longiglumis与四倍体Avena magna杂交获得,携带丰富的抗病基因,广泛应用于北美燕麦育种。基因组分析发现,部分Amagalon衍生品种中存在显著的结构变异,表现为6D染色体缺失与A.longiglumis来源的6A染色体替换。功能分析显示,这一染色体替换引发了明显的转录变化:6D染色体平均表达量显著低于6A,D亚基因组的表达抑制模式在6号染色体组中显著富集,表明该替换导致转录动态的重塑。尽管如此,这些携带替换的材料仍表现出优异的农艺性状,说明此类同源染色体间的交换在燕麦中具有良好的遗传稳定性和适应性。
八、结构变异对重组的影响
作者系统评估了SVs对燕麦育种中遗传重组的影响,对13个双亲群体中进行了基因分型与重组分析。研究发现多个杂交后代表现出显著的分离偏离及染色体内外的重组抑制,主要涉及染色体1A、1C、3C、4C和7D,这些区域与本研究及全球燕麦基因组多样性分析中确认的倒位与易位一致。进一步对两个半同胞交配群体的核型分析表明,一个群体在1A与1C染色体间存在伪连锁现象,由1A/1C互易易位的杂合型引起,同时7D染色体倒位杂合体也表现出重组抑制;另一个群体在3C染色体上出现重组受抑,与已知倒位相关。此外,作者还检测到其他潜在的结构变异杂合型,如1D与7C间的易位。
结语
本研究通过整合33份野生与栽培燕麦基因组,构建了高质量的燕麦泛基因组,并结合23份材料、6种组织的泛转录组图谱,系统揭示了燕麦基因组的结构与表达多样性。结果显示,C亚基因组整体表达水平较低;当A或D亚基因组拷贝缺失时,其同源基因往往能通过上调表达实现功能补偿,而C亚基因组的补偿能力显著较弱。研究还绘制了全基因组结构变异图谱,发现7D染色体的巨大着丝粒倒位与早抽穗性状密切相关,并揭示2A/2C同源臂易位在澳大利亚品种中广泛存在,可能与产量优势相关。此外,合成系Amagalon材料中出现6D被6A替代的独特事件。本研究为燕麦遗传改良与分子育种奠定了关键基础,推动燕麦功能基因解析与精准育种进程。
华命生物产品服务一览
华命生物目前已开通微信公众号、抖音、知乎、B站、小红书等线上平台,欢迎感兴趣的老师扫码关注了解更多内容!