GB精选| LTR-反转座子驱动棉花多倍体化
转座子(TEs)是植物基因组的重要组成部分,能够在基因组中扩和移动,推动遗传变异和基因组进化。长末端重复逆转录转座子(LTR-retrotransposons)是植物中最常见的转座子类型,它们通过复制机制增加基因组拷贝数,并可能导致基因组大小的扩张。在多倍化过程中,TE 的活动和扩增对基因组变化起着关键作用。棉属(Gossypium)是研究多倍化和作物改良的典型模型,TE 在其基因组扩张、物种分化和驯化过程中发挥了重要作用。
西班牙研究团队于2025年10月27日在国际知名期刊Genome Biology上发表了一篇题为“Differential LTR‑retrotransposon dynamics across polyploidization, speciation, domestication, and improvement of cotton (Gossypium)”的研究论文,探讨了在多倍体化、物种分化、驯化和育种过程中LTR-反转座子在棉花基因组中的动态变化, 为理解转座子驱动基因组演化与作物性状改良之间的联系提供了全新的基因组学视角。

一、LTR -反转录转座子谱系在四倍体棉花中有差异扩增
研究首先对八个陆地棉Gossypium hirsutum、十个海岛棉G. barbadense以及其双亲二倍体物种的基因组进行分析,发现四倍体棉中完整的长末端重复逆转录转座子的数量显著高于二倍体,尤其在 D亚基因组中密度几乎翻倍。插入时间分析表明,四倍体中的LTR元件更为年轻,显示出在多倍化及驯化过程中发生了近期的转座活化。进一步比较不同谱系发现,即使属于同一超家族,其动态变化亦存在显著差异。其中Copia家族的Tork和Ivana以及Gypsy家族的CRM谱系在多倍化后活性增强,而Gypsy的Athila、Ogre、Reina、Galadriel及Copia的Ale谱系则保持稳定。部分家族(如Copia Bianca、Ikeros、TAR)在所有基因组中拷贝数均较低。四倍体棉的长末端重复逆转录转座子呈现出谱系特异的动态演化,说明不同转座子在棉花基因组重塑与驯化过程中发挥了非均一的作用。
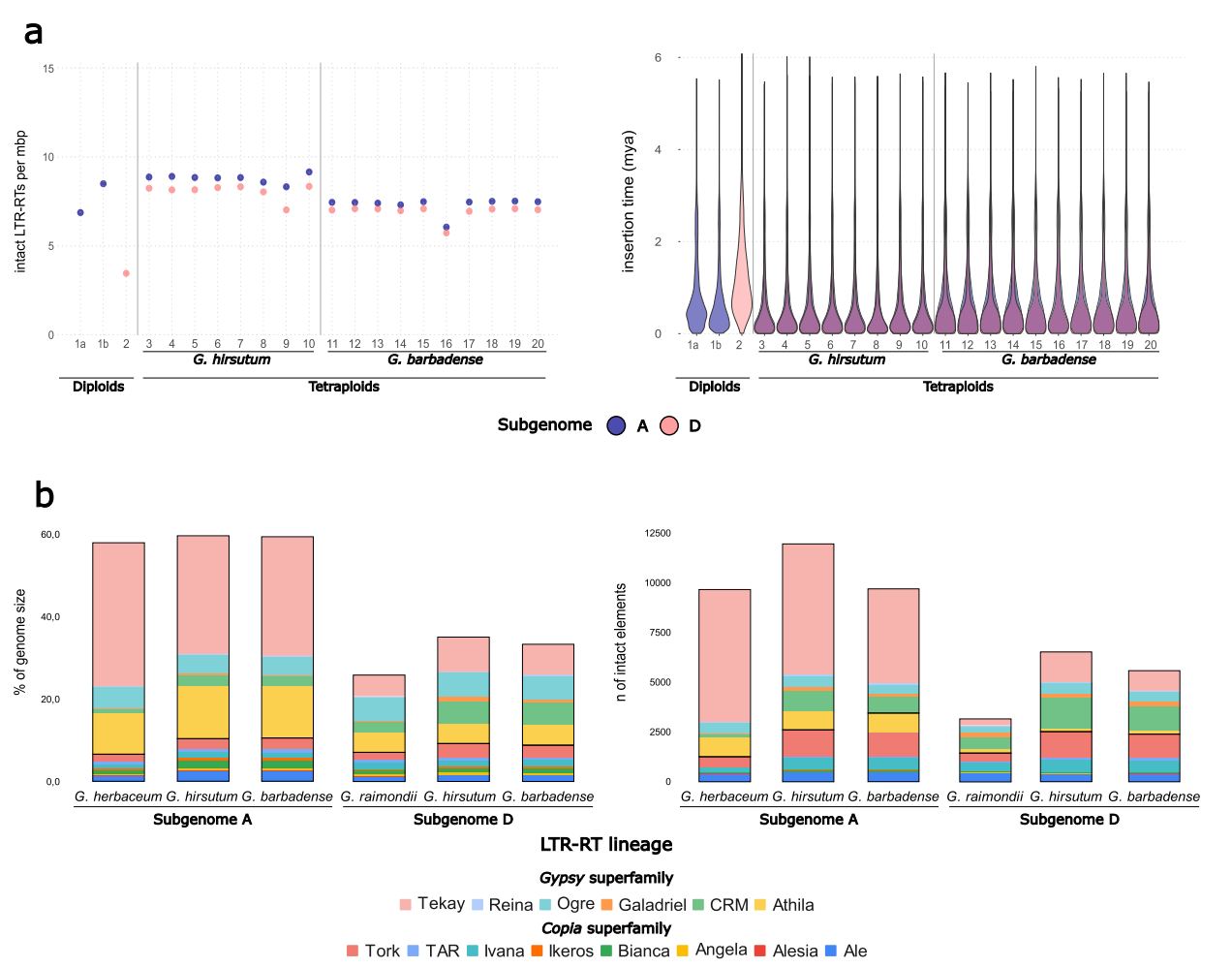
图1:二倍体和四倍体棉花中LTR-反转录转座子的亚基因组含量
二、LTR-逆转录转座子的清除速率在不同谱系、物种和亚基因组间存在差异
通过分析LTR内部重组形成的solo-LTR,作者发现陆地棉和海岛棉中超过90%的solo-LTR被靶位点重复序列(TSDs)包围,表明大部分为真实的solo-LTR。各家族间的消除动态差异明显:Tork元件在四倍体中表现出更高的反转座活性,而Athila家族的solo-LTR数量是完整元件的2-10倍,显示出较高的消除速率。Ogre家族在四倍体中保留了较多年轻插入,而 Ivana家族与Tork类似,活性较高、重组速率较低;Angela、Sire、TAR、Bianca与Ikeros家族的活性有限。Ale、Galadriel和Reina家族在四倍体与二倍体中活性相似,说明其在棉属近期进化中趋于静止。
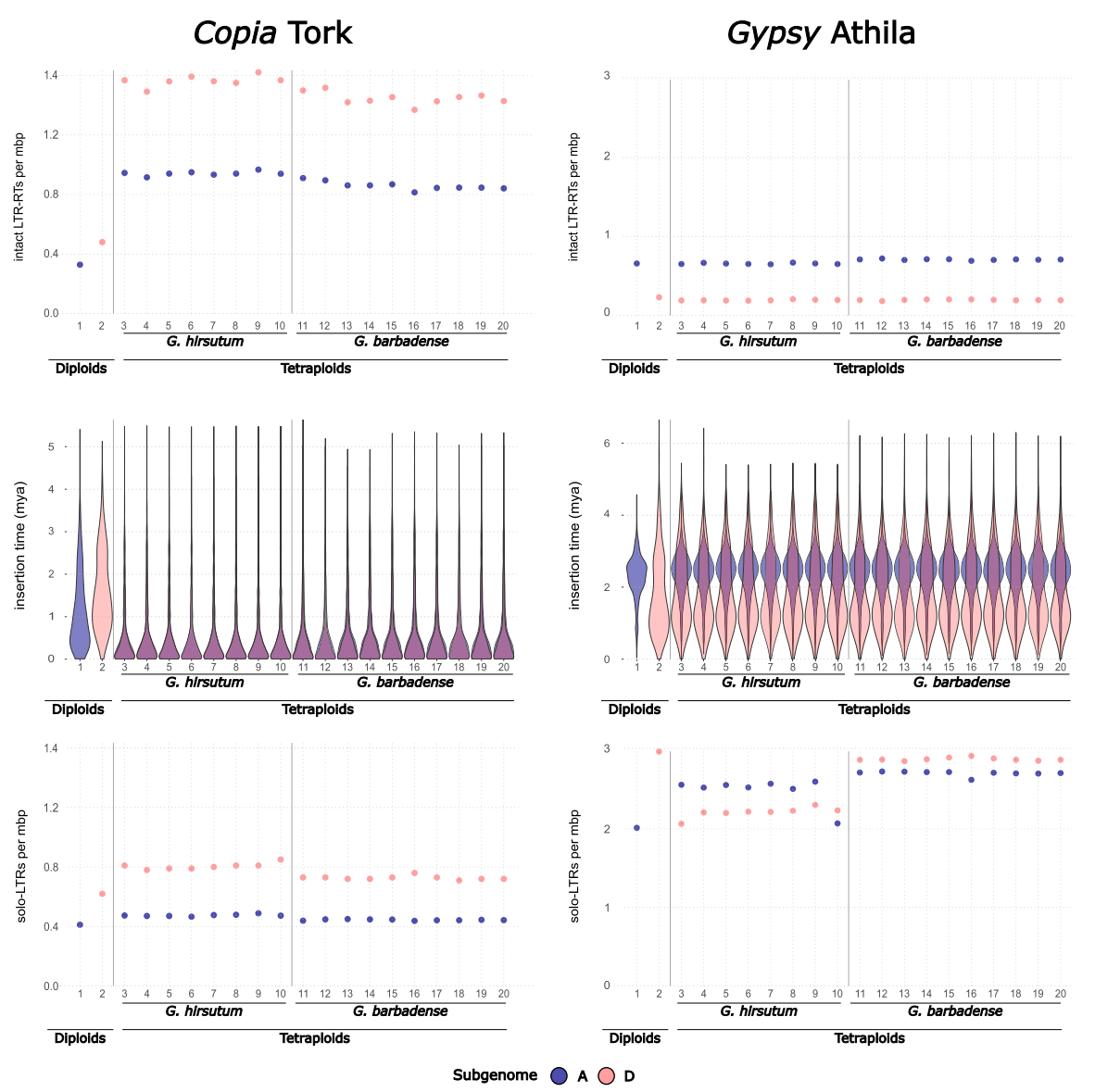
图2:完整的LTR-反转录转座子和单独的LTR Copia Tork和Gypsy Athila在二倍体和四倍体棉花中的亚基因组含量
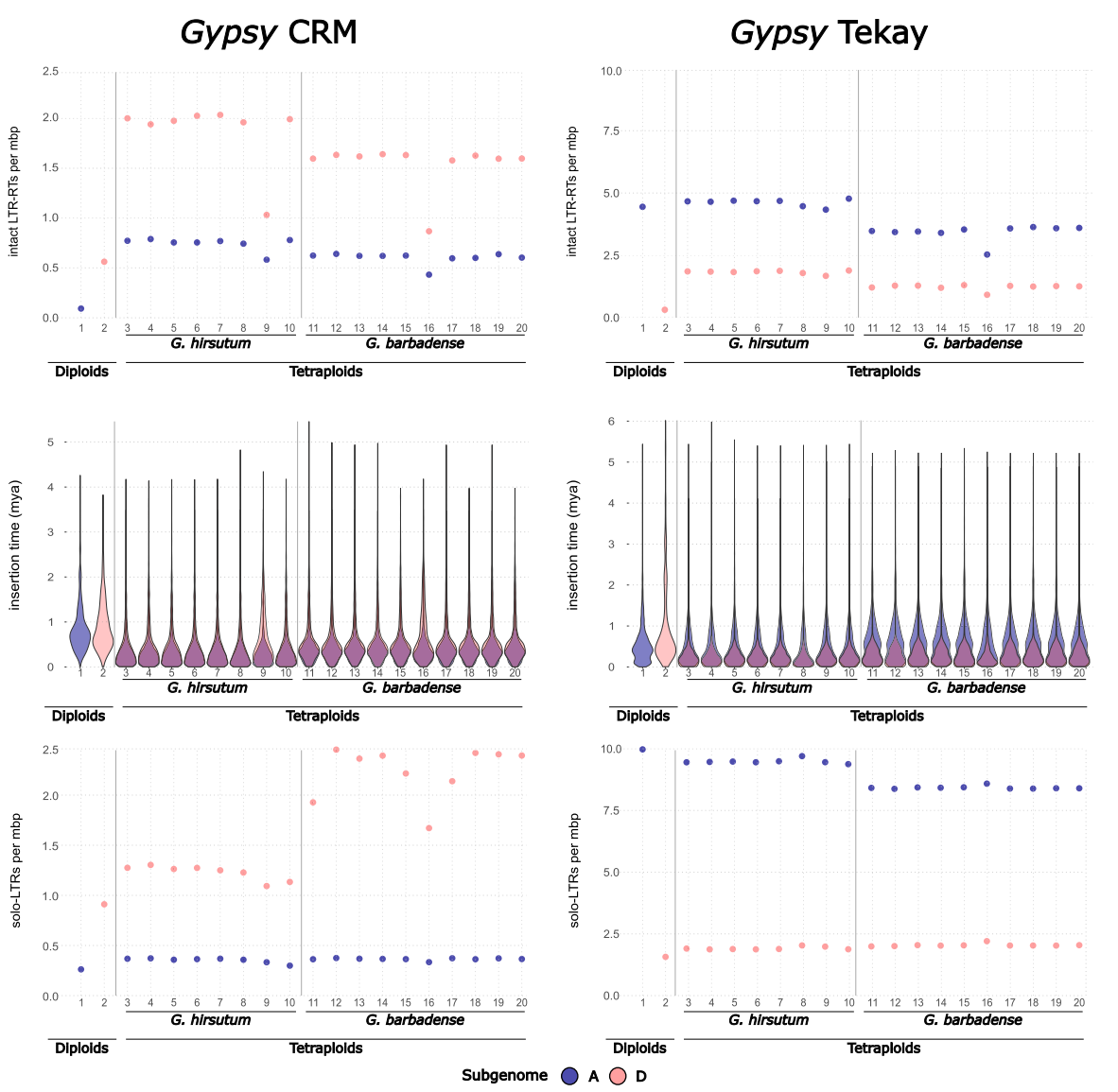
图3:完整的LTR-反转录转座子和单独的LTR Gypsy CRM和Gypsy Athila在二倍体和四倍体棉花中的亚基因组含量
三、CRM与Tekay的插入与清除塑造了四倍体棉的着丝粒结构
研究表明,CRM与Tekay元件的插入与消除在四倍体棉着丝粒的形成与重塑过程中发挥了关键作用。作者发现,CRM元件在多倍化后仍保持活跃转座,其拷贝数在陆地棉和海岛棉的A亚基因组中分别增加约8倍和6.5倍,而在D亚基因组中增幅较小。相比之下,Tekay元件主要在D亚基因组中扩增,而在A亚基因组中保持稳定或略有下降。solo-LTR分析显示,CRM在D亚基因组中具有更高的消除速率,尤其在海岛棉中最为显著;而Tekay的solo-LTR在A亚基因组中更为丰富,表明其重组效率较高且消除活跃。进一步分析显示,CRM完整元件显著富集于各染色体的着丝粒区域,而solo-LTR则沿染色体均匀分布,说明CRM元件在非重组区域中被优先保留。Tekay元件主要集中于近着丝粒区,其solo-LTR较少,可能在染色体臂区被清除。作者认为,CRM与Tekay在四倍体棉中表现出互补的扩增与清除模式,共同驱动了着丝粒结构的演化与基因组稳定性的维持。

图4:CRM和Tekay元件的染色体分布
四、少数LTR -反转录转座子谱系解释了种内多态性
研究进一步聚焦于四倍体棉花中LTR-反转座子在种内结构变异形成中的作用及其进化特征。作者通过构建陆地棉和海岛棉的泛基因组,对多个品种的基因组进行比对,识别出约6万处结构变异,其中约20%与LTR反转座子相关。通过检测插入多态性(TIPs),发现两种棉花均存在数千个完整LTR与solo-LTR插入,大部分具有靶位点重复(TSD),表明这些变异源于近期反转录转座事件。结果显示,尽管棉花基因组中存在众多LTR谱系,但仅有少数主导了种内多态性。一些谱系(如Ogre)在多倍化后短暂活跃,随后沉默;而CRM、Tekay、Ivana和Tork在物种分化后仍保持活性。不同谱系的多态性形式反映了其活性差异:CRM主要以solo-LTR形式存在,代表其经历了清除过程;Ivana和Tork多以完整LTR形式存在,显示持续的转座活动;Tekay则兼具两者,表现出插入与清除的动态平衡。空间分布上,CRM和Tekay集中于着丝粒区域,而Ivana和Tork广泛分布于基因附近,可能影响基因表达与表型变异。总体而言,棉花种内的基因组多样性主要由少数仍具活性的LTR反转座子谱系驱动,这些元件通过持续的转座与重组,推动了棉花基因组结构与功能的演化。
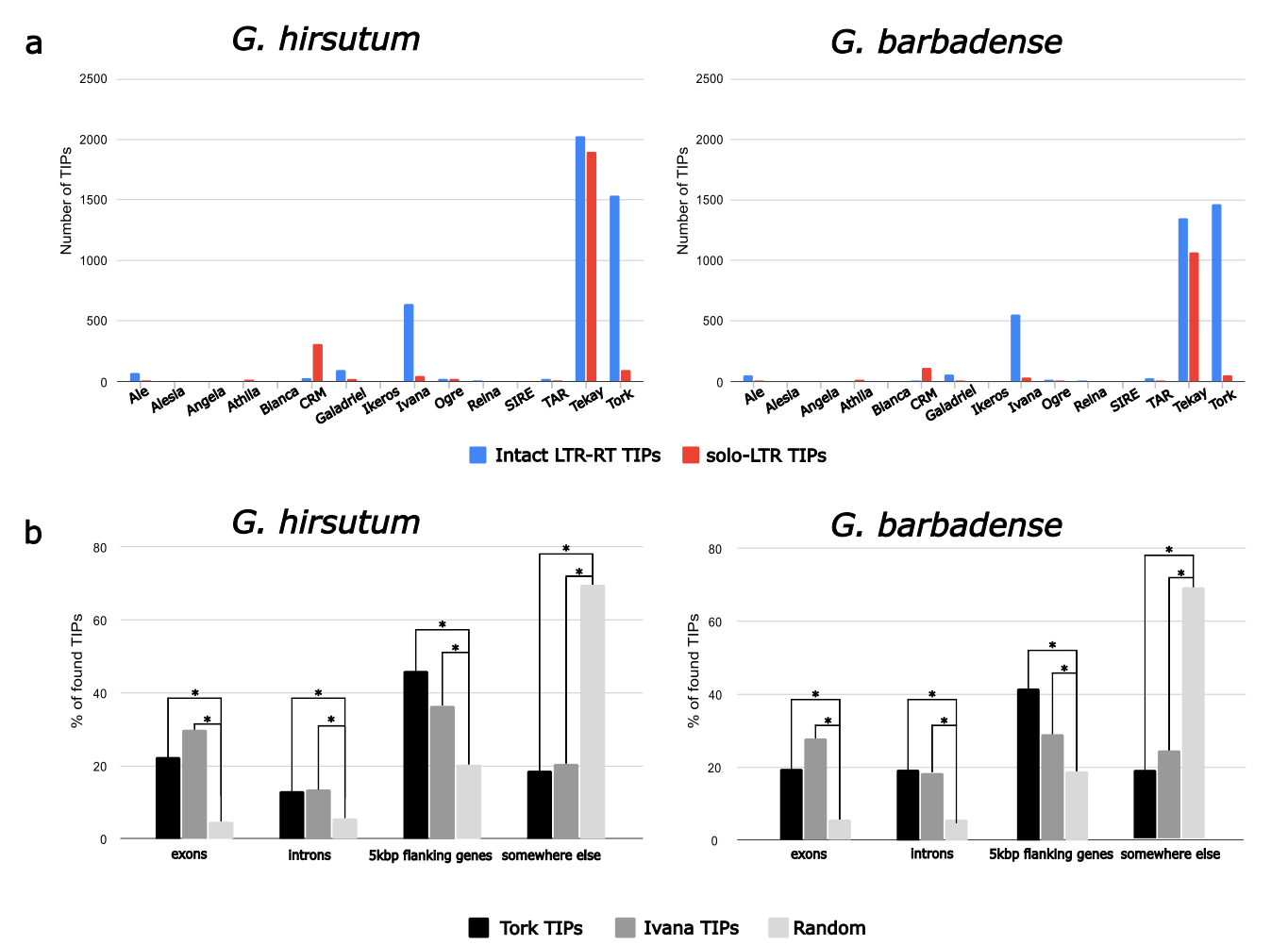
图5:LTR -反转录转座子和单独LTR插入的种内变异
五、LTR -反转录转座子TIPs对基因表达变异性的影响
在纤维发育过程中,作者对50个栽培品种的表达数据(8、12、16 DPA)进行TIP eQTL分析,研究结果表明共有187个TIP‑eQTL在三个发育阶段中显著关联,其中58个(31%)在所有阶段中出现,50个(26%)在两个阶段中出现。功能富集分析显示,这些关联基因在“响应生长素”和“生长素激活信号通路”以及“脂肪酸代谢与延伸”等类别中显著富集,与细胞壁沉积和纤维延长密切相关。大多数TIP的插入效应为负面(80%),特别是在编码区内的插入;然而,也有少数插入在基因上下游近端区域导致基因激活。进一步分析表明,多态插入主要来源于Tork谱系和Ivana谱系,还包括Gypsy元素的solo-LTR TIPs。这些结果表明,LTR-反转录转座子的近距离插入可影响纤维发育相关基因的表达,进而对该物种的基因表达变异性产生重要影响。
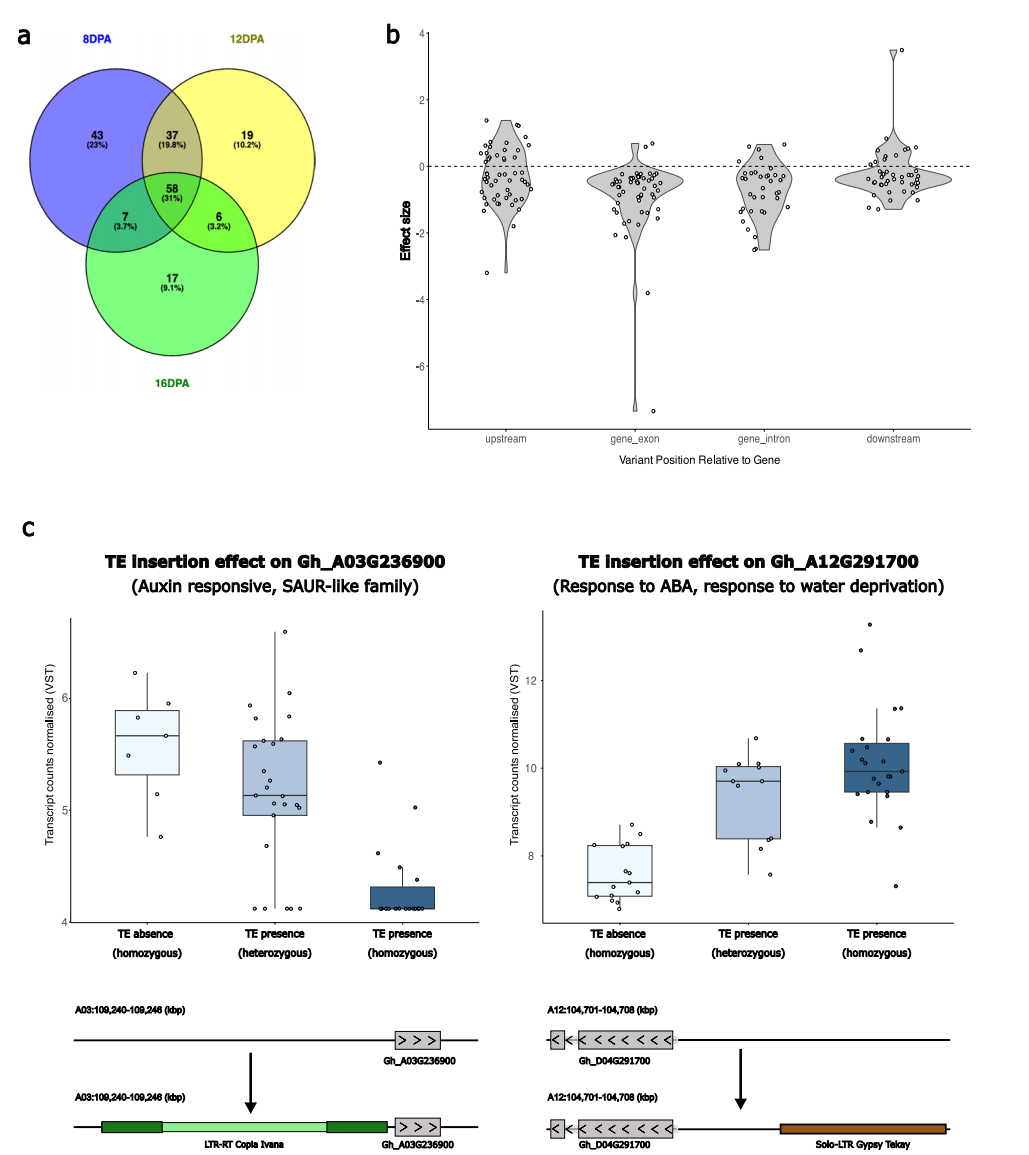
图6:在50个陆地棉品种群体中发现TIP-eQTL
六、LTR -反转录转座子TIPs对棉花驯化和育种的影响
对陆地棉和海岛棉群体的TIPs分析表明,LTR-反转座子介导的基因组变异在棉花驯化与改良过程中可能发挥了重要作用。通过对野生型、地方品种及现代栽培品种的泛基因组比对与基因分型,研究发现TIP数据可有效区分不同进化阶段的群体,揭示在驯化过程中转座活动及拷贝保留/丢失模式存在显著差异,尤其在陆地棉中表现出明显的遗传多样性收缩特征。进一步的群体分化指数(PBS)分析显示,一部分TIP在不同群体间频率差异显著,其邻近基因可能受到正向或负向选择。功能富集结果表明,这些TIP附近的基因在“生长素反应”通路中显著聚集,涉及多种SAUR样生长素响应蛋白及吲哚乙酸诱导蛋白基因,说明其在棉花的生长发育与适应性进化中具有潜在功能意义。类似的富集现象也在海岛棉中观察到,说明与生长素相关的基因变异可能在两种栽培棉的驯化和育种过程中被共同选中。然而,这些差异亦可能部分来源于群体结构或种间基因渗入的历史背景,其具体作用机制尚需进一步验证。
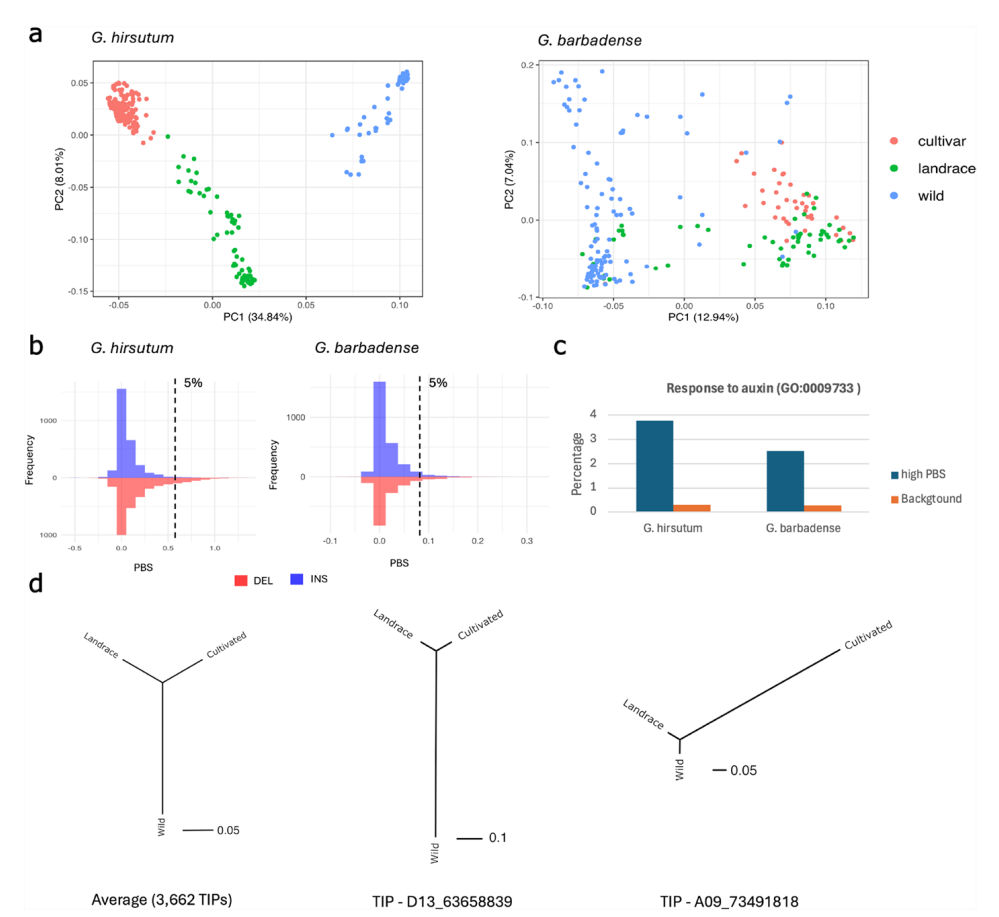
图7:伴随陆地棉和海岛棉驯化与育种过程的TIP频率变化
结语
本研究探讨了LTR-反转座子在棉花基因组中的动态变化,特别是在多倍体化、物种分化、驯化和育种过程中的作用。研究发现,棉花的多倍体物种相较于二倍体祖先,LTR-反转座子在D亚基因组中的数量显著增加,且不同的转座子谱系在基因组中表现出不同的扩增和沉默模式。通过分析陆地棉和海岛棉品种的TIP频率,揭示了这些转座子插入在驯化和育种过程中的潜在作用。PBS分析表明,与生长素响应相关的TIP插入可能在育种过程中受到选择。进一步的TIP eQTL分析显示,LTR-反转座子插入可能影响纤维发育相关基因的表达,表明这些插入在棉花驯化过程中对纤维性状产生了重要影响。本研究揭示了LTR-反转座子在棉花基因组演化、物种驯化和育种过程中发挥的重要作用,特别是在塑造基因组变异、影响基因表达以及改良经济性状方面的潜力。
华命生物产品服务一览
华命生物目前已开通微信公众号、抖音、知乎、B站、小红书等线上平台,欢迎感兴趣的老师扫码关注了解更多内容!