NC重磅 | 野燕麦near-T2T基因组揭示除草剂抗性位点
野燕麦Avena fatua是与栽培燕麦 Avena sativa和不实野生燕麦A. sterilis密切相关的六倍体杂草,在全球农田中广泛分布,被认为是危害最严重的杂草之一。它具备极强的环境适应性和表型可塑性,能够耐受多种非生物和生物胁迫,并表现出显著的除草剂抗性。尽管其入侵性对农业生产造成严重损失,野燕麦仍蕴藏着丰富的抗病抗逆遗传资源。
河北大学杜会龙教授及其研究团队于2025年11月7日在国际著名期刊Nature Communications上发表了题为“Reference genome and population genomic analyses reveal insight into herbicide tolerance in Avena fatua L.”的研究论文,构建了野燕麦高质量参考基因组及群体变异图谱,揭示其与栽培燕麦的遗传关系,并鉴定出位于4D染色体、包含扩增GST基因簇的关键除草剂抗性位点,为培育高抗性燕麦品种提供了重要基因资源。

一、野燕麦的高质量基因组组装、注释和比较基因组学
研究共获得306.43 Gb的PacBio HiFi、317.73 Gb的ONT以及196.64 Gb的Hi-C数据,构建了一个总长度为10.98 Gb、contig N50达473.48 Mb的高质量基因组组装。结果显示,99.79%的序列成功锚定到21条染色体,基因组完整性达到98.5%,整体质量优于目前已发布的所有燕麦基因组。基因组注释结果表明,重复序列占组装基因组的88.38%,其中主要为反转录转座子,Gypsy型LTR转座子占总转座元件长度的35.17%。
系统发育分析显示,野燕麦与带壳燕麦(Sang)和裸燕麦(Sanfensan)的分化时间约为0.79-1.13百万年前,是栽培燕麦的最近外群。结构变异比较发现,野燕麦基因组中存在大量特有的大片段倒位,主要分布在染色体4A和4C上,这些结构在不实野生燕麦和栽培燕麦中缺失。进一步的比较基因组分析表明,野燕麦相较于栽培燕麦扩增了472个基因家族,涉及胁迫响应与解毒相关的GRAS、GST和UGT等家族。其中GRAS家族在A亚基因组中显著扩增,而GST家族扩增最为明显,特别是染色体4D上Tau类GST基因的特异性增加。RNA-seq分析显示这些GST基因在根和叶中高表达,参与解毒代谢和除草剂耐受。
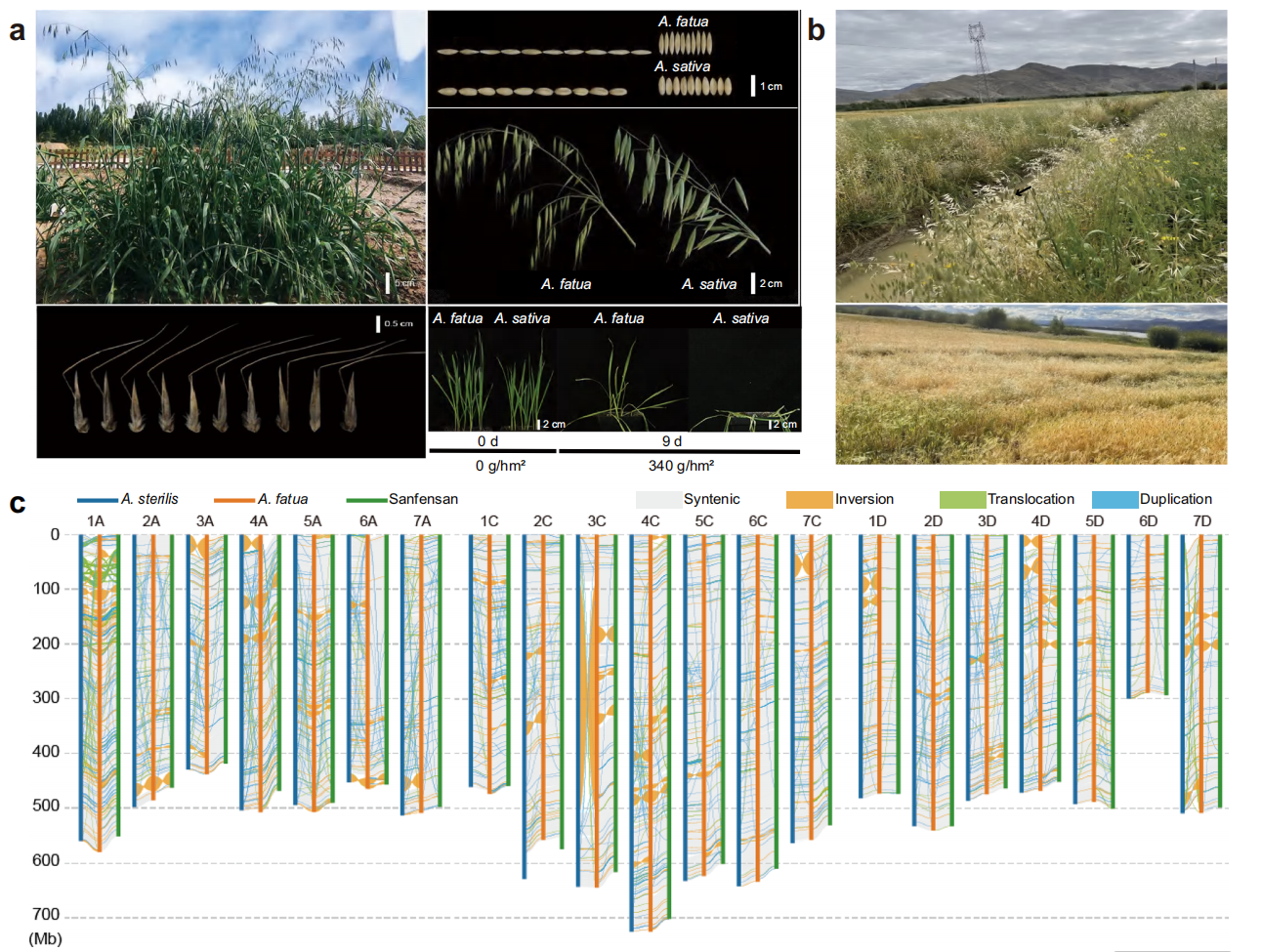
图1:野燕麦的表型特征及W1004材料基因组组装概览
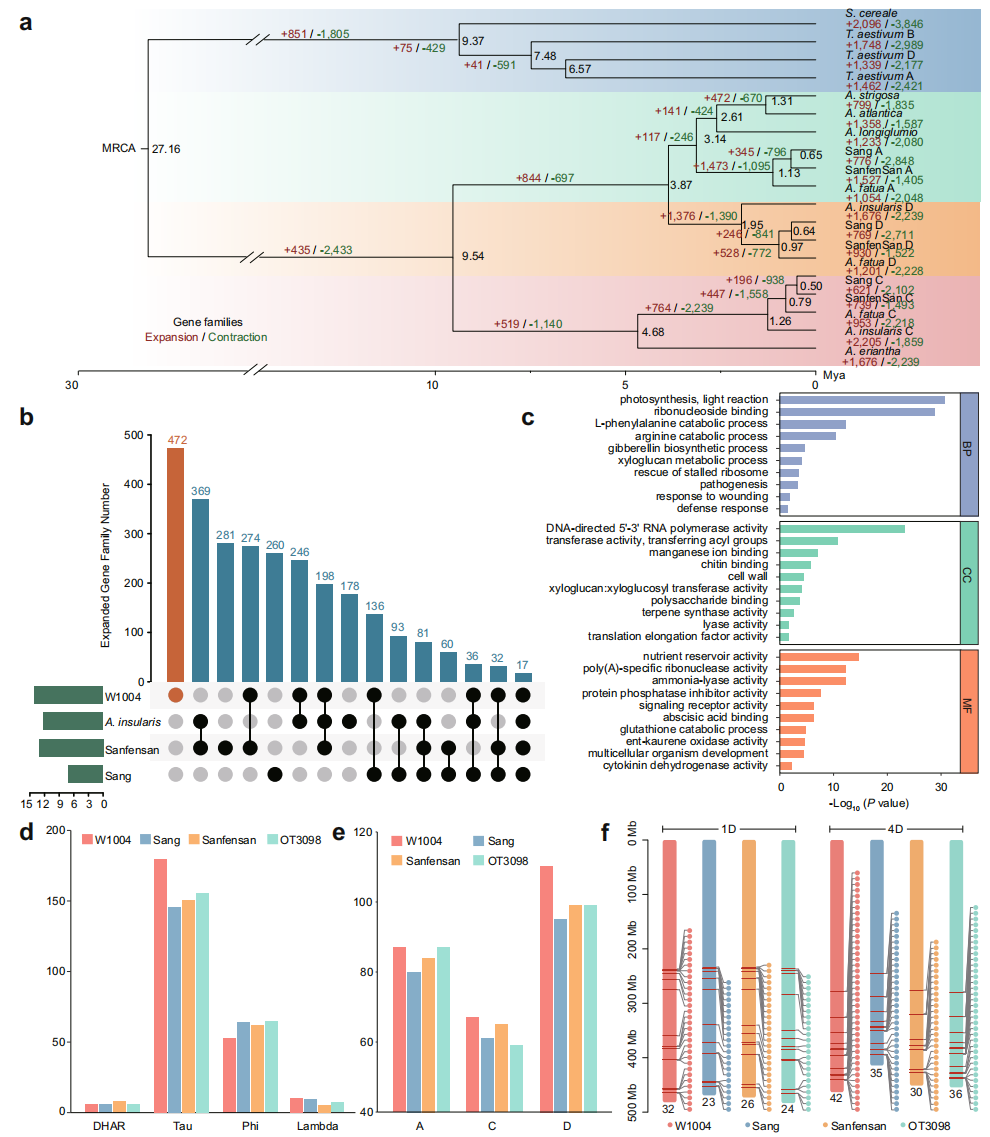
图 2:燕麦属基因组系统发育树及野燕麦W1004株系的基因家族分析
二、野燕麦、栽培燕麦与不实野生燕麦的遗传关系及进化历史
作者利用来自中国北方青藏高原、新疆和张家口地区的443份野燕麦样本,以及全球范围内288份栽培燕麦的全基因组重测序数据,并结合既有的37份野生燕麦基因组,对燕麦群体进行了系统遗传分析,共获得7600余万个高质量SNP位点。主成分分析与群体结构分析结果一致表明,不实野生燕麦作为栽培燕麦的祖先类群,与栽培燕麦显著分化;栽培燕麦进一步分为带壳燕麦与裸燕麦两支。野燕麦在遗传上与裸燕麦最为接近,二者间遗传分化程度最低,D统计结果支持其可能存在基因交流或直接进化关系。遗传多样性分析显示,裸燕麦的核苷酸多样性最低,而野燕麦最高,其中青藏高原群体的多样性尤为丰富,且呈现随海拔降低而递减的趋势。连锁不平衡分析进一步揭示,栽培群体在驯化过程中经历了显著的遗传瓶颈。此外,在野燕麦中检测到少量源自不实野生燕麦的遗传成分,说明历史上可能发生过杂交事件。
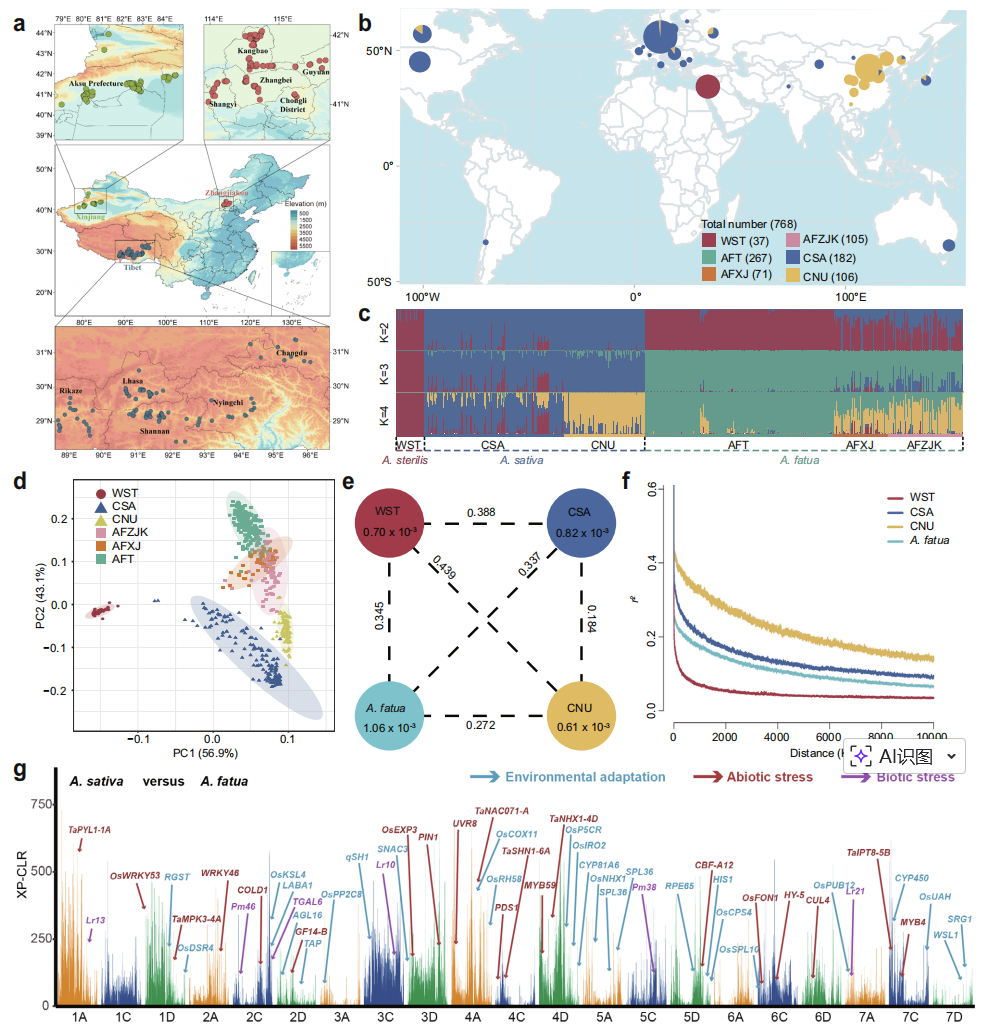
图3:野燕麦、栽培燕麦和不实野燕麦的群体遗传学分析
三、野燕麦环境适应性的基因组选择特征
研究进一步探讨了野燕麦对极端环境广泛适应性的遗传机制,将其与近缘的栽培燕麦的群体基因组进行对比,采用六种方法(包括基因组覆盖度差异、结构变异频率、跨群体延伸单倍型纯合性XP-EHH、复合似然比XP-CLR、固定指数Fₛₜ、以及核苷酸多样性比值π)识别出其在野燕麦群体中可能受到选择的基因组区域。结果表明,这些方法共同识别出约1.11-1.71Gb的候选区域,覆盖12896-22051个基因,其中2417个基因被至少五种方法检测到,表明野燕麦和栽培燕麦间存在显著遗传分化。
功能富集分析显示,这批候选基因主要集中在两大类:一是“胁迫响应”(包括氧化应激、激素信号等),二是“生长/发育”相关。有105个基因为已知耐逆基因的同源(涵盖重金属、盐、热、寒、旱、抗病、抗除草剂等),另有51个基因与农艺性状(如产量/品质、生长/发育、不育、脱粒)同源。应激相关基因在两物种之间序列相似性较低,表明其在环境适应性方面分化更为显著。

图4: 野燕麦和栽培燕麦遗传差异区域的全基因组扫描
四、野燕麦除草剂响应机制的多组学分析
为阐明野燕麦的除草剂耐受机制,作者整合转录组与表观基因组分析,系统探讨了其在氟苯草酯胁迫下的动态响应。RNA-seq结果显示,除草剂处理共诱导了23833个差异表达基因(DEGs),其中叶片与根部分别有一组基因在不同时间点持续响应。这些基因显著富集于氨基酸代谢、激素信号、谷胱甘肽代谢及DNA损伤修复等防御相关通路。同时,作者通过ATAC-seq在叶组织中鉴定出大量除草剂诱导的染色质开放区域(DARs)。通过整合分析,发现了1908个基因的表达变化与染色质可及性改变直接相关,并将其划分为四个具有不同表达模式的簇(G1-G4)。各簇基因受不同的转录因子(如SBP、bZIP、NAC等)调控,并呈现出时序性的功能转换:处理前高表达基因主要参与基础代谢与激素合成;而处理后激活的基因则转向钙信号、蛋白磷酸化及氧化应激响应。值得注意的是,谷胱甘肽S-转移酶(GST)基因簇在处理后被显著诱导,突出其在解毒和耐药过程中的核心作用。

图5:野燕麦材料 W1004 在除草剂处理下的转录组与染色质可及性分析
五、qGST4D 是野燕麦除草剂耐受性的关键位点
为进一步解析野燕麦除草剂耐受的基因组基础,作者以氟苯草酯处理后的存活率为表型指标,进行了全基因组关联分析。发现其除草剂耐受性主要受位于第4D染色体末端的qGST4D位点调控。该位点(4D:442-444Mb)与基因扩增及选择清扫区域一致,包含一簇谷胱甘肽S-转移酶(GST)基因,其中11个基因对除草剂处理产生响应。研究进一步确认该区域内扩增基因4Dg0135144在野生燕麦中分布频率高达96.74%,而在栽培燕麦中仅为53.41%。qRT-PCR与ATAC-seq结果显示,除草剂处理显著诱导其在根和叶中高表达,并增强了染色质开放性。通过TRV介导的基因沉默实验,4Dg0135144表达量下调导致植株存活率、CAT酶活性、糖含量和叶绿素显著下降;相反,过表达植株表现出更强的除草剂抗性与更高的CAT活性。综合结果表明,4Dg0135144是qGST4D区域内介导除草剂耐受的关键基因。
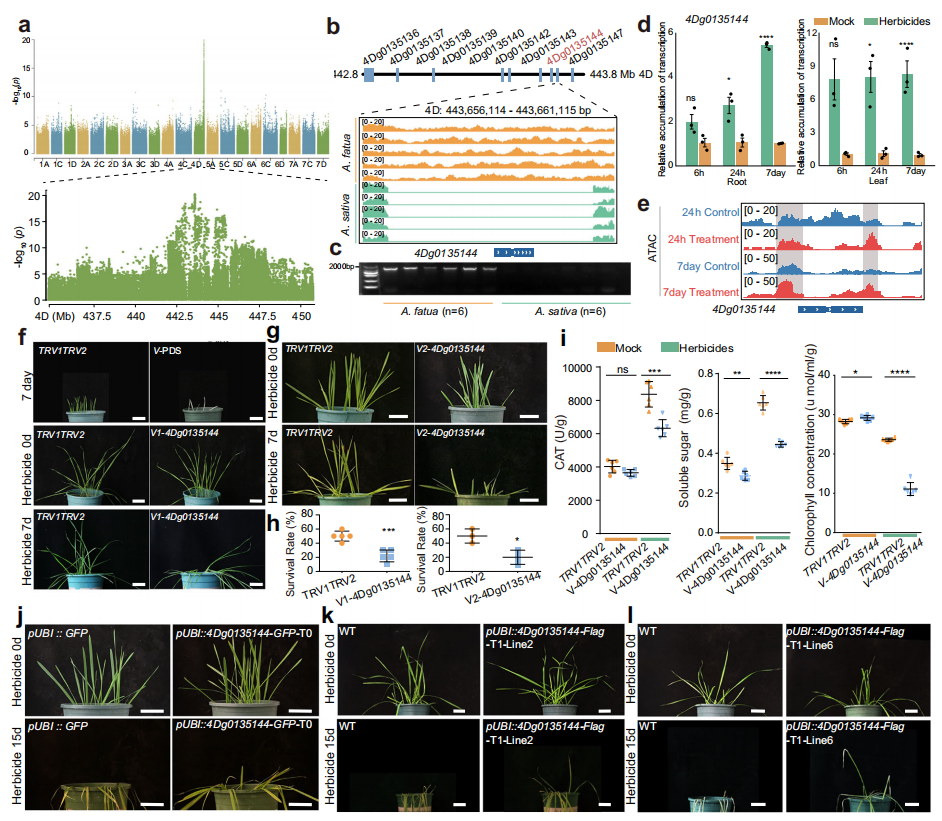
图6:qGST4D是野燕麦除草剂耐受性的关键位点
结语
本研究成功构建了六倍体野燕麦10.98Gb参考基因组,并对768份野生及栽培燕麦材料开展了群体重测序分析,系统解析了野燕麦与栽培燕麦(尤其是去壳燕麦)之间的遗传联系与演化历史。在全基因组水平,研究识别出多个显著富集胁迫相关基因的分化区域,并重点关注到位于4D染色体末端的一个谷胱甘肽S-转移酶(GST)基因扩增簇(qGST4D)。通过整合多组学分析与全基因组关联分析,研究进一步验证了该簇中一个扩增基因(4Dg0135144)可显著增强除草剂耐受性:敲低该基因导致植株存活率下降,而过表达则显著提升抗性。该成果揭示了野燕麦在环境适应与代谢性除草剂耐受方面的遗传基础,并为未来培育耐逆、抗除草剂的栽培燕麦品种提供了关键基因资源与分子靶标。
华命生物产品服务一览
华命生物目前已开通微信公众号、抖音、知乎、B站、小红书等线上平台,欢迎感兴趣的老师扫码关注了解更多内容!