MP精选 | 全球首个燕麦T2T基因组
华命生物目前已成功完成50+物种的T2T基因组组装,物种涵盖动物、植物、昆虫及同源和异源多倍体等疑难物种,已有多个合作项目在顶级期刊发表和接收,欢迎有需要的老师垂询。联系方式:18371456025。
普通燕麦(Avena sativa,2n=6x=42,AACCDD)作为全球重要的粮食与饲料作物,对贫瘠边缘环境具有极强适应力,但受限于庞大基因组(约11Gb)、高重复序列占比及多倍体复杂性,其基因组研究与分子育种进展显著滞后于其他主粮作物。
2025年2月,河北大学杜会龙教授团队在国际权威期刊Molecular Plant上在线发表了题为A gap-free complete genome assembly of oat and OatOmics, a multi-omics database 的论文,首次构建了六倍体燕麦的端粒到端粒(T2T)基因组,这是目前首个超过10Gb的T2T基因组组装,为复杂基因组解析树立了新标杆。

一、基因组组装与质量评估
通过整合462.59Gb的PacBio HiFi数据、706.01Gb的 ONT超长数据和384.54Gb Hi-C数据,作者组装获得了包含全部21个着丝粒和42个端粒的10.99Gb基因组(Marv-T2T)。分析发现较OT3098和Sanfensan,分别新增325.51Mb/302.75Mb序列及5,541/4,577个新注释基因。
作者通过多种基因组质量评估了Marv-T2T的高质量组装。
◎序列比对率:NGS/HiFi/Hi-C数据>99.96%,ONT数据99.23%
◎基因完整性:BUSCO评估值99.60%(A/C/D亚基因组分别达98.1%/89.2%/95.6%)
Merqury质量值:64.27
◎LTR index:17.88(OT3098仅17.42,Sanfensan 13.93,Sang 4.35)
◎重复序列:占基因组88.40%(9.71Gb)
◎实验验证:通过PCR确认所有填补间隙准确性

图1:Marv-T2T组装和质量评估
二、复杂区域和C亚基因组解析
Marv-T2T为解析复杂基因组区域结构提供了前所未有的研究机遇,通过结合CENH3 ChIP-seq数据,作者在Marv-T2T、Sang、OT3098和Sanfensan中鉴定了着丝粒区域。并发现各染色体着丝粒长度存在显著差异(5A染色体最长:10.31Mb;3A最短:5.32Mb),并且与Gypsy转座子拷贝数呈正相关,表明Gypsy扩增驱动了燕麦着丝粒的进化扩张。
而Marv-T2T中CENH3富集区(平均7.53Mb)显著大于其他基因组chr2C的预测值,凸显了T2T对着丝粒区域解析的重要性。
另外发现着丝粒区富含玉米着丝粒反转录转座子(CRM),但C亚基因组CRM含量显著低于A/D亚基因组。除C亚基因组外,着丝粒区LTR-RT插入时间均显著年轻于非着丝粒区。
进一步分析发现C亚基因组着丝粒区LTR-RT插入时间均显著早于A/D亚基因组,提示C与A/D谱系分化后经历了独特进化。另外C亚基因组含有特有的着丝粒串联重复序列Cen48和Cen87。
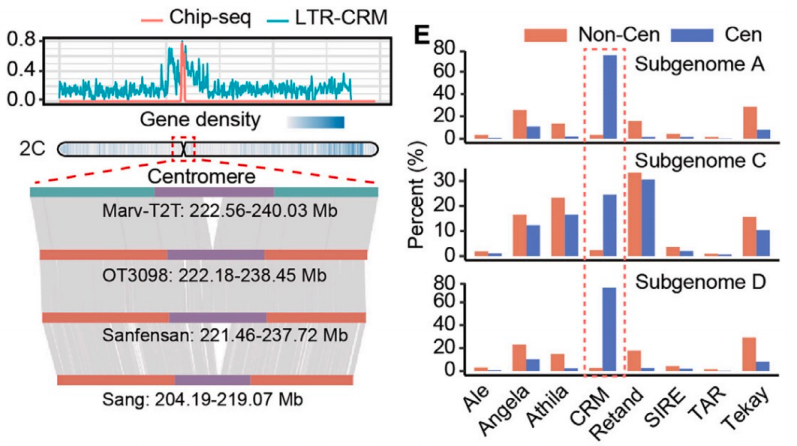
图二:2C染色体着丝粒和ACD亚基因组TE超家族
三、OatOmics:燕麦多组学整合研究平台
作者构建了整合基因组学、多组学与表型组学资源的综合性平台OatOmics(http://www.oatomics.com/)。该数据库包含以下核心模块:
3.1基因组数据和注释
◇收录12个已测序燕麦属物种基因组
◇采用统一流程重新注释,确保数据可比性
◇支持共线性分析与基因检索
3.2 基因组变异
◇包含25个国家/地区1079份种质的31,541,757个高质量SNP
◇揭示不同群体遗传关系与分化
3.3 表型数据库
◇收录株高、旗叶长宽、鲜/干重、茎粗、分蘖数等8个关键性状
◇整合蛋白质含量、油酸、精氨酸含量及主穗粒重相关候选位点
3.4 核心种质库
◇基于基因型-表型-地理多样性筛选254份代表性种质
◇覆盖原群体97.7%的SNP基因型
3.5 基因表达谱
◇获取拔节期转录组数据构建表达变异图谱
◇支持表达数量性状位点(eQTL)分析与调控基因挖掘
3.6 分析工具集
◇集成JBrowse2基因组浏览器、共线性检索、BLAST比对
◇提供KEGG/GO富集、多序列比对、热图绘制
◇内置全基因组关联分析(GWAS)、差异表达分析、ORF预测等工具
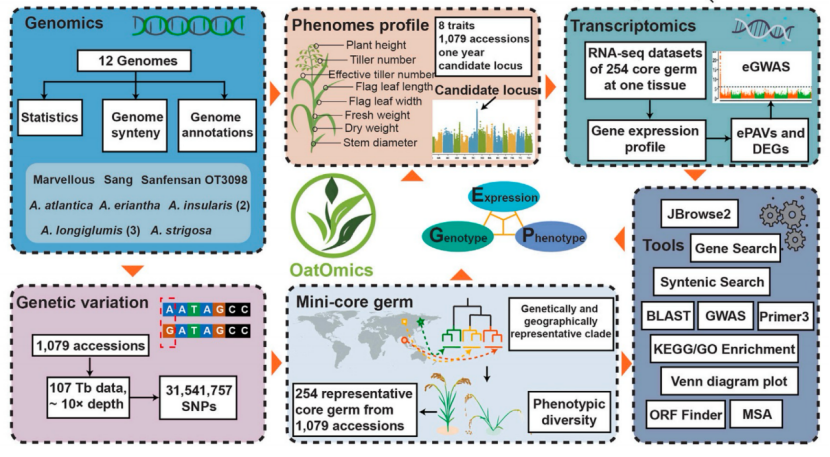
图3:OatOmics数据库
研究结论
本研究成功构建了首个超过10 Gb的燕麦端粒到端粒无间隙基因组(Marv-T2T),为解析燕麦基因组的"暗物质"及进化机制提供了前所未有的研究基础。同时,作者开发了OatOmics多组学平台,整合了全球1079份燕麦种质的基因组、转录组、变异组及表型组数据,并提供系列分析工具,助力高效利用这些资源。
华命生物产品服务一览

华命生物目前已开通微信公众号、抖音、知乎、B站、小红书等线上平台,欢迎感兴趣的老师扫码关注了解更多内容!


