NP精选 | 野生蔷薇基因组大数据的精准育种策略创新
华命生物目前已成功完成50+物种的T2T基因组组装,物种涵盖动物、植物、昆虫及同源和异源多倍体等疑难物种,已有多个合作项目在顶级期刊发表和接收,欢迎有需要的老师垂询。联系方式:18371456025。
蔷薇属植物的栽培与驯化历程映射出不同文化间的交流与审美观念的演变,最终形成了当今最具观赏价值的植物类群。然而,栽培玫瑰狭窄的遗传基础制约了其进一步改良,野生蔷薇物种蕴含着丰富的遗传多样性,但其开发利用长期受分类学争议的阻碍。
2025年4月4日,来自北京林业大学的于超教授团队在著名期刊《Nature Plants》上发表了题为“Phenotypic and genomic signatures across wild Rosa species open new horizons for modern rose breeding”的研究论文,本研究通过构建波斯蔷薇(Rosa persica)单倍型T2T基因组,对大量蔷薇属样本进行了系统发育和群体基因组分析,全面解析了蔷薇属植物的驯化历程,为未来利用野生资源进行再驯化及创新育种工作奠定了坚实基础。

一、波斯蔷薇T2T基因组组装
本研究完成了波斯蔷薇(Rosa persica,2n=2x=14)的单倍型T2T基因组组装。通过整合86.56 Gb(247×)PacBio HiFi测序数据与55.37 Gb Hi-C数据,通过多轮组装和手工调整,最终获得0 Gap的单倍型基因组,其中Hap 1总长为364.44 Mb,Hap 2为363.41 Mb。并且鉴定出28个端粒(每个单倍型14个),并检测到两个单倍型的14个着丝粒区域(长度1.1–4.3 Mb,平均2.2 Mb)。
二、蔷薇属种质重测序与系统发育解析
为全面解析蔷薇属内系统发育关系,本研究整合了涵盖该属全谱系的种质资源。通过多年野外考察结合表型测定、标本比对及系统发育证据,经严谨鉴定,最终确定205份蔷薇属种质,涵盖该属全部3个亚属和10个组,对上述材料进行全基因组重测序,整合10份已发表种质数据,最终获得涵盖215份蔷薇属种质的高质量数据集。数据比对至波斯蔷薇Hap1参考基因组进行变异检测,筛选高质量SNP用于后续分析。
基于707个单拷贝核基因获得6,048个保守SNP,构建了蔷薇属全属系统发育树。该树显示单性蔷薇亚属(subg. Hulthemia,进化枝A)处于基部位置。主亚属(subg. Rosa)呈现并系群结构,分化为两大高支持率进化枝(枝B与C)。
基于测序数据组装205份种质的叶绿体基因组,利用注释编码序列重建了蔷薇属叶绿体系统发育树。叶绿体系统树与核基因组树总体趋势一致,多数组内种质聚为单系群,但部分样本也存在显著拓扑差异。
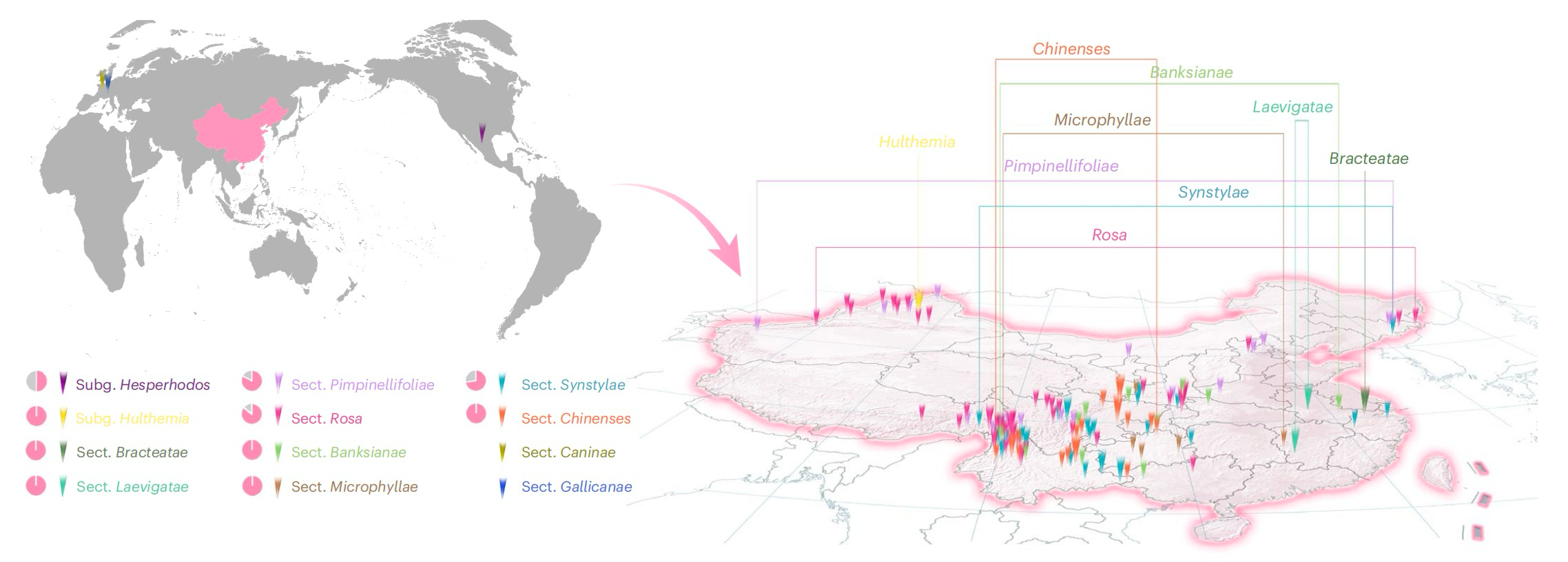
图1:核心蔷薇属的地理分布情况

图2:基于215份蔷薇种质资源的707个单拷贝核基因保守性SNP的系统发育关系
三、蔷薇属关键性状祖先状态重建
通过最大简约法与最大似然法对五类形态性状进行祖先状态重建,揭示了蔷薇属重要性状的演化轨迹。两种方法均显示:单生单瓣黄花且无花瓣斑点为蔷薇属祖先性状。小叶数目在系统发育中经历多次转变,祖先状态最可能为7枚。花瓣数目分析表明,单瓣花为祖先状态。花序结构演化方面,单生花为原始特征,向伞房或复合花序的转变可能发生两次:首次出现于木香组分支,第二次发生于合萼组与栽培玫瑰进化枝。木香组与合萼组间存在潜在基因交流,推测该性状可能从木香组随后传递至近期演化类群。花色演化分析支持黄花为蔷薇属祖先状态。长期人工选择使重瓣、艳丽花色等性状成为育种核心目标,导致现代玫瑰与祖先特征产生显著分化,同时揭示波斯蔷薇等原始种质对现代玫瑰的遗传贡献相对有限。

图3:最大简约法与最大似然法重建蔷薇属关键性状祖先状态
四、蔷薇属群体结构揭示种内分化格局
基于215份样本的PCA显示,主成分PC1(51.1%)有效区分早期分化种质与近期演化类群。单性蔷薇亚属、西野蔷薇亚属、刺玫组与蔷薇组的早期分化种质聚为一簇,而合萼组、月季组与栽培玫瑰等近期分化类群形成独立聚类。
利用ADMIXTURE进行解析,最优群体数K=9。群体遗传结构与系统发育树高度吻合,并支持叶绿体与核基因组树的拓扑差异。栽培玫瑰遗传组分几乎完全源自月季组与合萼组,进一步证实这两个组群是现代栽培玫瑰的主要供体来源。
根据群体结构、PCA与系统发育结果,将215份种质划分为早期分化群(群组1)与近期分化群(群组2),其中群组2进一步分为野生种与栽培玫瑰。连锁不平衡和π值计算表明,玫瑰驯化过程中的人工选择导致群体遗传多样性降低,而近期分化群组内野生种质仍保留较高遗传变异,为蔷薇属资源保护与育种创新提供了重要理论依据。

图4:蔷薇属群体结构分析
五、蔷薇属起源与种群历史重建
基于地理分布特征,蔷薇属在中国呈现西北与西南两大分化中心。TreeMix与遗传结构分析支持双中心起源假说。作者进一步分析推测蔷薇组起源于刺玫组,合萼组可能起源于木香组,而月季组为合萼组的衍生类群。
通过有效种群数量动态分析,发现蔷薇属约600万年前经历首次种群瓶颈,该时期正值中新世-上新世过渡阶段,气候剧变导致种群规模锐减,驱动组间分化。第二次种群瓶颈发生于末次盛冰期,全球低温干旱导致蔷薇属种群持续萎缩。
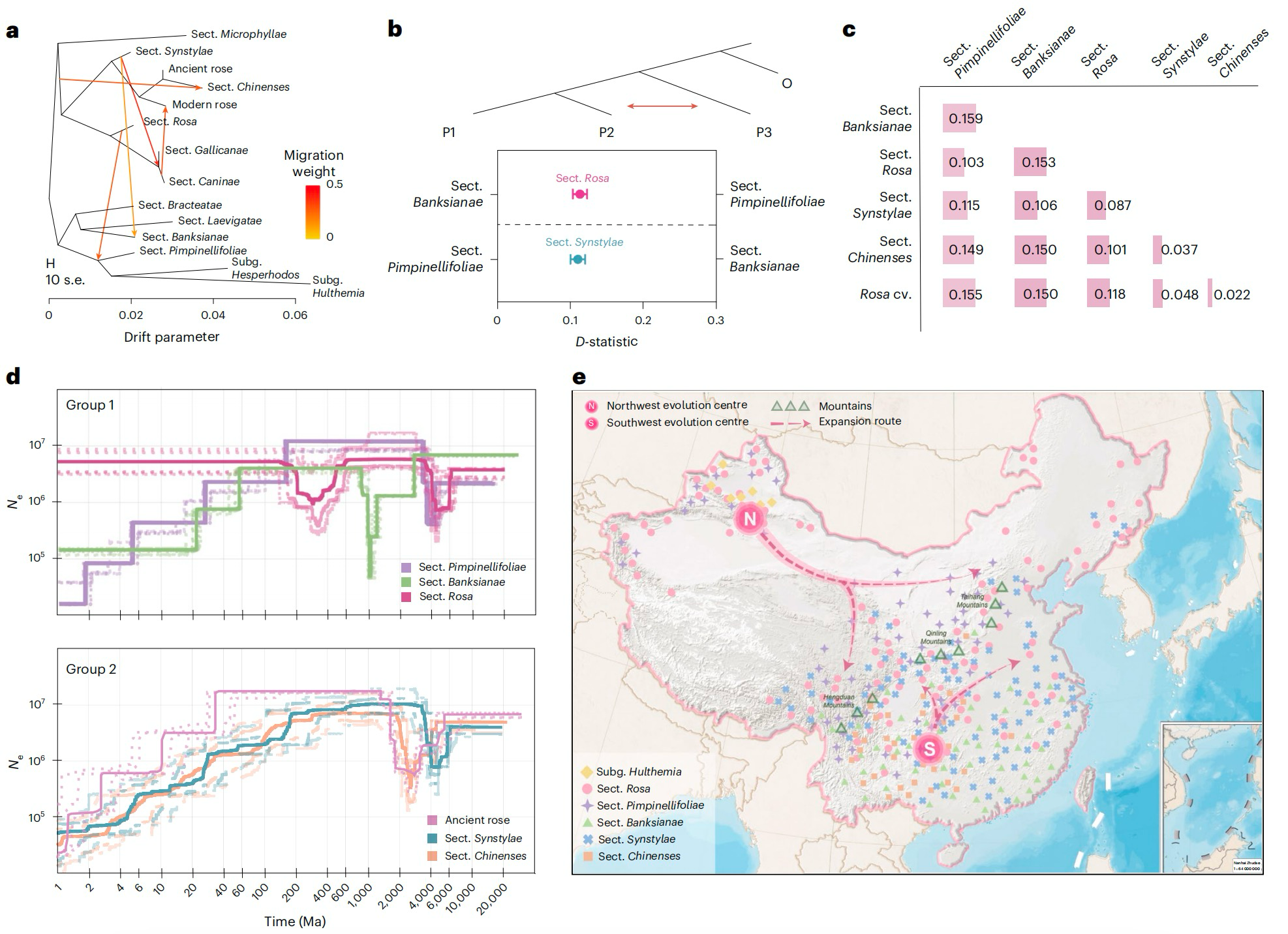
图5:蔷薇属种群历史变化解析
六、蔷薇属驯化性状基因组足迹鉴定
作者进一步通过选择清除分析,系统解析蔷薇属演化与驯化过程中的关键基因组区域。包括花色、香味、连续开花、花器官形态和抗病性基因等,发现MYB基因在花色演化中的关键调控作用,通过结合GWAS分析揭示了花瓣数量的关键QTL等性状相关区域。

图6:蔷薇属群体全基因组鉴定与选择清除分析
研究结论
本研究对大量蔷薇属样本进行了基于全基因组测序的分析。通过利用波斯蔷薇的单倍型T2T基因组组装数据,构建了全面的蔷薇属系统发育框架。这项研究通过揭示蔷薇属的植物学起源、系统发育关系及演化历史,为野生蔷薇资源的开发利用奠定了坚实基础,助力了现代月季的再驯化与革命性育种工作。
华命生物产品服务一览

华命生物目前已开通微信公众号、抖音、知乎、B站、小红书等线上平台,欢迎感兴趣的老师扫码关注了解更多内容!


