基因组里程碑丨中国科学家NG发表六倍体小麦T2T基因组
华命生物目前已成功完成50+物种的T2T基因组组装,物种涵盖动物、植物、昆虫及同源和异源多倍体等疑难物种,已有多个合作项目在顶级期刊发表和接收,欢迎有需要的老师垂询。联系方式:18371456025。
普通小麦(Triticum aestivum L.)是全球广泛种植的农作物,普通小麦基因组大小约15 Gb,包含三个亚基因组,其中重复序列占比超过85%,这使其成为植物基因组测序的重大挑战。2018年,国际小麦基因组测序联盟(IWGSC)首次发布了中国春(CS)的参考基因组,随着测序技术的进步,已有20多个普通小麦基因组完成组装。但这些基因组仍存在大量的区域未组装完全,且着丝粒和端粒未被完整解析。
2025年4月7日,潍坊现代农业山东省实验室/北京大学现代农业研究院邓兴旺、何航和李博生团队在国际著名期刊《Nature Genetics》上发表题为“A telomere-to-telomere genome assembly coupled with multi-omic data provides insights into the evolution of hexaploid bread wheat”的突破性成果文章,通过整合长读长测序与综合分析,成功组装了异源六倍体普通小麦(中国春)的T2T参考基因组CS-IAAS,标志着小麦基因组研究的里程碑。CS-IAAS具有高度的完整性和精确性,为小麦基因组序列、结构、基因表达与功能特征的深入研究提供了宝贵资源,并为未来作物改良提供了关键见解。

一、中国春小麦T2T基因组组装
为构建完整的小麦基因组,作者整合了PacBio HiFi测序(3.8Tb,250×)、ONT ultra-long测序(>100 kb,1.8Tb,120×)、Hi-C(100×)和Bionano(200×)数据,并开发了半自动化的T2T参考序列组装流程SPART,通过初步组装和缺口填补,最终获得的CS-IAAS基因组全长14.51 Gb,21条染色体均为0 gap,contig N50高达723.78 Mb。完整包含42个端粒和21个着丝粒。相比CS RefSeq v2.1,CS-IAAS新增565.66 Mb的序列。

图1:六倍体小麦T2T基因组CS-IAAS组装情况
二、小麦亚基因组重排与演化
作者系统比较了二倍体(单粒小麦、拟斯卑尔脱山羊草、粗山羊草)、四倍体(二粒小麦、硬粒小麦)和六倍体(中国春与Kariega)的共线性关系。研究发现四倍化过程中存在高达223种染色体重排,并首次通过高质量基因组序列精确定位4A染色体易位事件,而六倍化过程中未检测到显著易位,但发现23个保守的染色体倒位(总长度为518 Mbp)倒位区域包含2,083个基因。
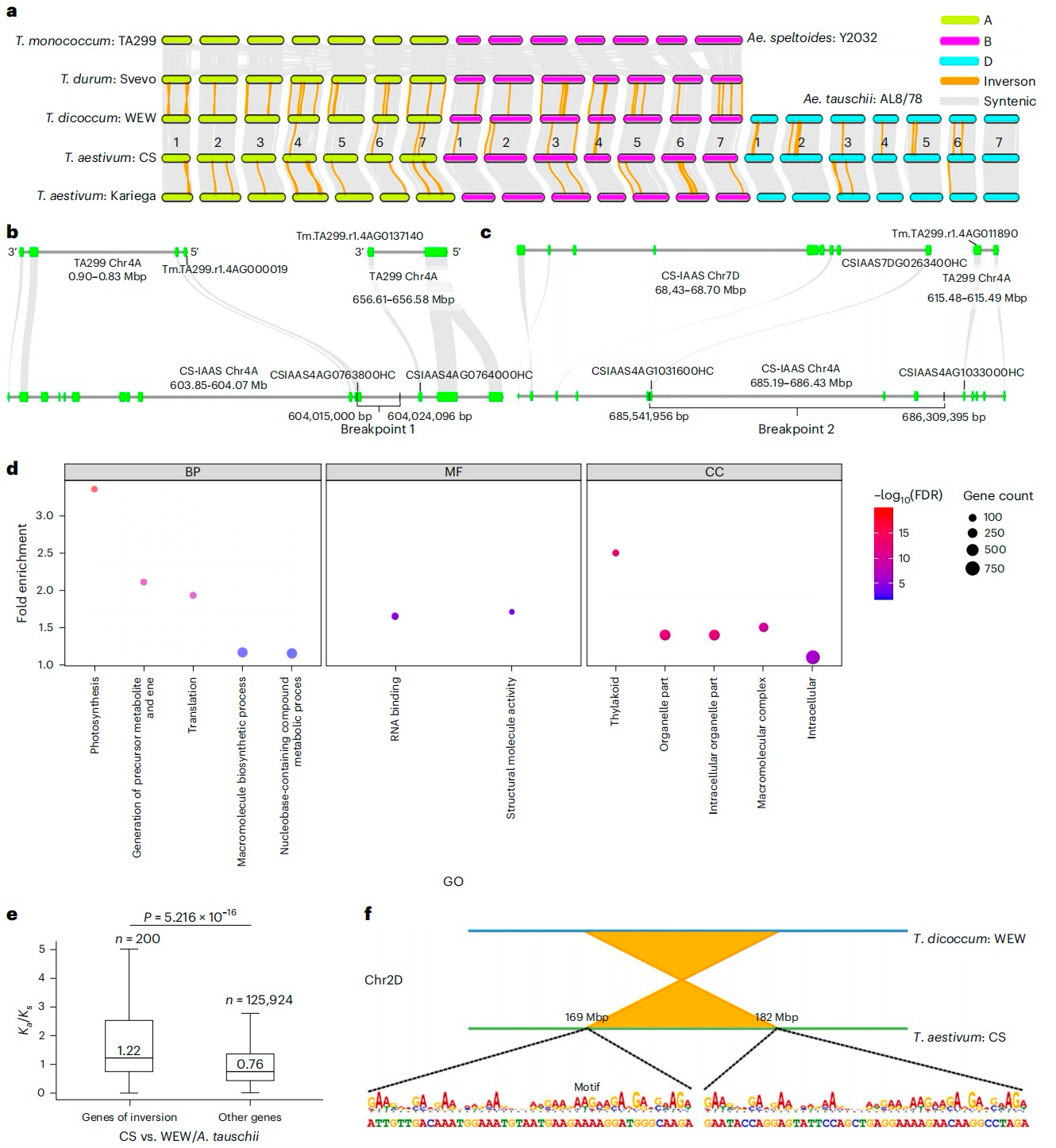
图2:小麦亚基因组重排与六倍化演化
三、亚基因组特异性rDNA结构解析
本研究完整组装50.86 Mbp rDNA阵列,鉴定5,611个完整rDNA单元,1A/5D染色体主要含有短串联重复,1B/6B染色体分别解析1,991和3,302个单元,8个功能分区中,IGS间隔区与26S区长度变异最大,而3'-ETS序列仅存在于B/D亚基因组。
另外通过植物端粒重复序列(TTTAGGG,拟南芥型序列),作者在全部21条染色体的两端均鉴定出了完整的端粒结构。端粒的长度范围为4,718 bp至32,663 bp,拟南芥型序列占小麦端粒序列的75%,而脊椎动物型TTAGGG序列占16%。
四、转座元件(TE)与片段重复(SD)的进化特征
基于T2T CS-IAAS基因组完整组装,作者首次系统解析了此前难以研究的重复序列区域。在CS-IAAS基因组中鉴定出12.34 Gb的TE序列,并新发现了212.62 Mb TE序列。研究发现两个近期扩张的DNA转座子亚家族,扩张发生于六倍化之后。并且D亚基因组中年轻转座元件的比例显著高于A、B亚基因组,提示其近期可能发生基因渗入。
通过Biser分析,64.83%的CS-IAAS基因组被鉴定为SD序列。共识别出25,540个初级SD,归类为7,754个SD集合,其中1,156个为核心重复集合。A和B亚基因组的SD集合数量显著高于D亚基因组,且各亚基因组存在特异性SD集合,仅510个集合为三者共享。
进一步分析发现,TE相关基因对的Ks值更低,且表达水平普遍低于SD区域基因,可能与TE周边高甲基化水平有关。此外,TE插入对基因表达平衡具有调控作用:A/B/D亚基因组中低表达基因周围TE含量显著高于优势表达或平衡表达基因。这些发现表明,TE插入通过降低基因表达增强了多倍体基因组的表达可塑性。

图3:CS-IAAS揭示六倍体小麦TE的组成和分布情况
五、亚基因组着丝粒结构特征
CS-IAAS基因组组装为小麦着丝粒结构研究提供了全新视角,研究精确重构了总长192.7 Mbp的小麦着丝粒区域,其中TE占比超过94%。分析发现:Gypsy家族CRM元件在着丝粒显著富集,Retand家族被CENH3特异性靶向,是首个被报道具有着丝粒活性的Gypsy亚家族,Athila与Tekay家族在不同染色体着丝粒中呈现动态变异。
六倍体A亚基因组与二倍体祖先相比长度出现了显著扩增,CRM数量也出现了激增,着丝粒区域共鉴定271个高置信度基因(HC基因),其中D亚基因组含136个HC基因(占比50%),这些发现揭示了着丝粒作为多倍化过程中亚基因组分化的关键区域,其通过转座元件驱动的动态重塑促进了小麦基因组演化。

图5:六倍体小麦着丝粒序列组成解析
六、高精度基因注释与全长转录组分析
作者利用全长转录组测序技术(Iso-Seq)构建了面包小麦的全长转录组图谱,涵盖小麦不同发育阶段的14种组织及愈伤组织。共鉴定出163,329个非冗余全长转录本。通过整合167个样本的公开RNA-Seq数据、本研究新生成的全长转录本及已报道的FLcDNA数据库。由此构建的综合性基因注释包含141,035个高置信度(HC)蛋白质编码基因模型,19,680个基因以单拷贝形式分布于各亚基因组(1:1:1),占HC基因总数的41.86%。同时,在A、B、D亚基因组中分别鉴定出18,819、22,098和17,874个亚基因组特异性基因。
基于高精度基因注释和全长转录组数据,作者构建了小麦可变剪接事件资源库。共鉴定出29,123个AS事件,保留内含子(RI)型为最主要的剪接类型。通过Iso-Seq技术检测到12,927个高置信度(HC)基因参与AS事件,这些基因相较于所有HC基因具有更长的序列和更高的表达水平。在19,862个组织特异性AS事件中,愈伤组织占比最高。而在1,044组具有同源关系的HCs基因中,所有成员均存在AS事件。其中349组呈现AS事件的不平衡分布,68组在某一亚基因组中显著富集,而281组则在某一亚基因组中显著缺失,揭示了亚基因组间剪接调控的复杂性。

图6:全长转录组解析基因结构注释和AS事件
七、六倍体面包小麦蛋白质组草图解析
基于高精度CS-IAAS基因组,作者最后采用先进质谱技术对三种组织进行深度蛋白质组分析。共鉴定出高置信度(HC)基因对应的36,701个蛋白质组,其中29,902个HC蛋白质具有独特肽段支持。蛋白质组数据为HC基因的开放阅读框(ORF)边界注释提供了重要支持,这些发现验证了本研究所构建蛋白质组数据的精确性及基因注释体系的完整性。
华命生物产品服务一览

华命生物目前已开通微信公众号、抖音、知乎、B站、小红书等线上平台,欢迎感兴趣的老师扫码关注了解更多内容!


