动物T2T基因组
一、产品介绍
自2020年7月Nature上首次发表人类基因组完整的X染色体序列以来,T2T联盟致力于人类基因组的完善及修正。2022年3月31日,端粒到端粒(T2T)联盟公布了首个完整、无间隙的人类基因组序列,首次揭示了高度相同的节段重复基因组区域及其在人类基因组中的变异。开启了基因组组装的T2T时代。
T2T基因组是基因组组装的终极目标,是指所有染色体达到端粒到端粒(Telomere-to-Telomere)的水平的gap-free基因组,它能矫正原来版本基因组中的组装错误,完善原有版本的基因组,有助于对基因组中高重复序列区域或高重复结构进行深入研究,为着丝粒区域及未知高重复区域的变异特征的研究提供契机,是功能基因组学和分子育种的基础。
二、测序策略
Pacbio HIFI + Nanopore ultral long + HIC
三、分析亮点
基因组denovo组装中的gap填补;端粒和着丝粒的鉴定

四、T2T基因组能解决哪些问题
五、为什么选择T2T基因组
01 填补空白传统基因组测序技术无法覆盖复杂区域(如端粒、着丝粒和重复序列),T2T基因组首次解决了这一问题。
02 新发现在为测序区域中发现了新基因和功能元件,为基因组研究开辟了新方向。
03 进化研究为物种基因组进化和多样性研究提供了全新工具。
04 医学突破为理解遗传疾病、基因组变异和个性化医疗提供了更完整的参考。
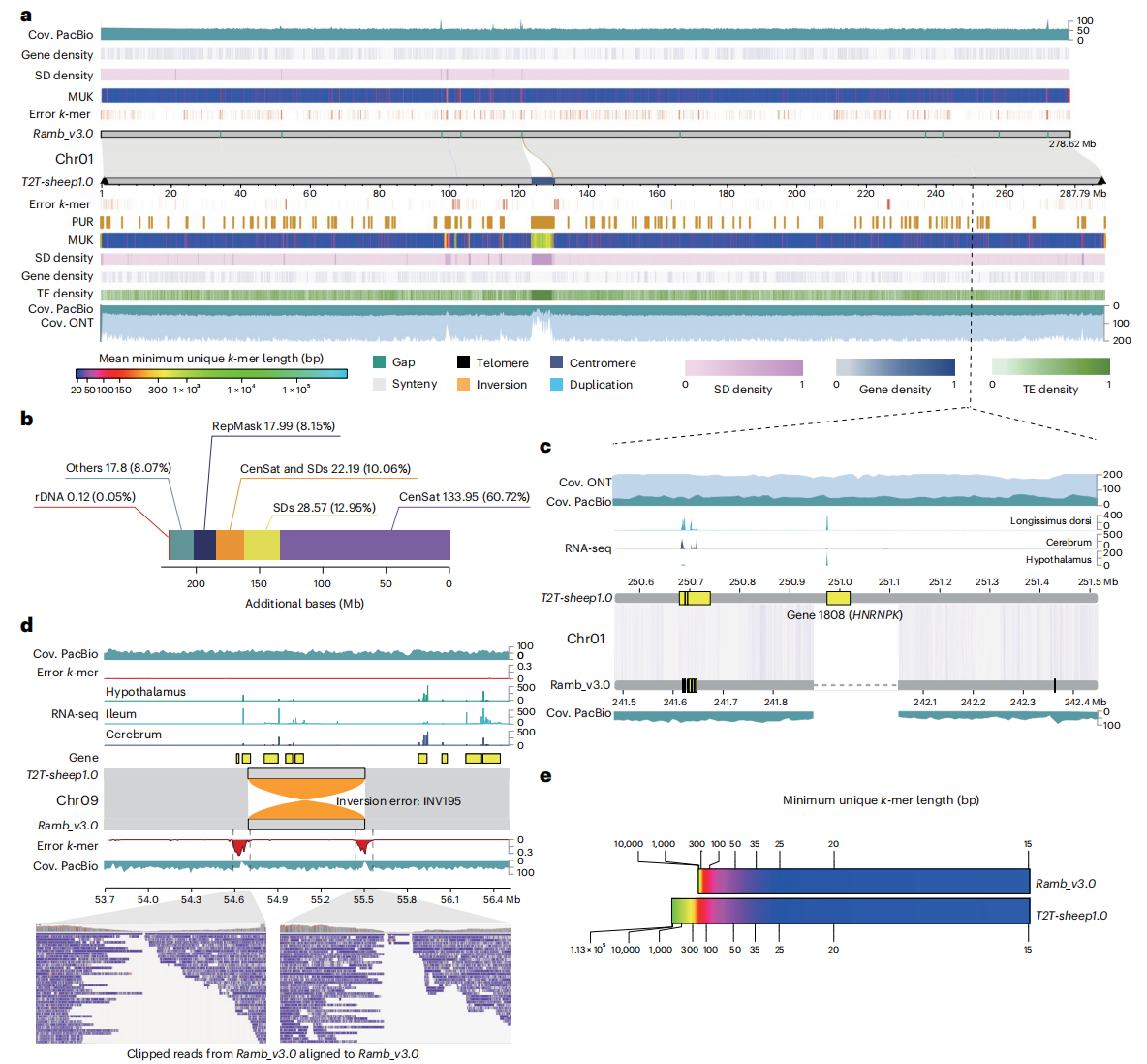
六、文献案例:中国农大李孟华团队发表首个绵羊T2T参考基因组
【文章题目】Telomere-to-telomere sheep genome assembly identifies variants associated with wool fineness
【发表期刊】Nature Genetics

【主要研究内容】该研究首次完成了中国著名的高繁殖力品种——湖羊(HU3095)的端粒到端粒无间隙基因组组装(T2T-sheep1.0),其中包括完整的Y染色体(T2T-sheep1.0-chrY)和着丝粒区域的组装。除此之外,该研究报道了T2T水平的单倍型基因组组装,即父本基因组T2T-sheep1.0P和母本基因组T2T-sheep1.0M。基于T2T-sheep1.0参考基因组,该研究对着丝粒、端粒结构以及当前参考基因组中未解析的区域(PURs)进行了深入解析。同时,结合三代PacBio测序数据以及全球范围内野生和家养绵羊的全基因组重测序数据,该研究进行了变异检测和群体分析,结果表明,与当前的绵羊参考基因组Ramb_v3.0相比,T2T-sheep1.0在比对性能和变异检测方面表现出显著优势,有助于鉴定新的与驯化和毛用性状相关的选择信号和候选基因。绵羊端粒到端粒的完整基因组组装将为绵羊遗传育种相关的研究提供新的见解。