T2T泛基因组:58个阿联酋人完整基因组图谱
人类参考基因组是基因组学和生物医学研究的重要基础,但当前版本(包括HPRC的47例双倍体组装及T2T的CHM13单倍体基因组)仍未能充分反映全球人群的遗传多样性,导致对某些群体的基因变异分析存在偏差。虽然中国、非洲等地已开展了泛基因组研究,但在中东地区,尤其是阿拉伯半岛人群的基因组特征仍缺乏深入研究。这些人群具有独特的遗传背景、较高的近亲婚配比例以及复杂的历史迁徙过程,这使得现有参考基因组难以准确描述他们的遗传结构。
哈利法大学的Habiba Alsafar研究员及其科研团队近日发表一篇题目为“The Emirati T2T-Level Pangenome: A Graph of 58 Complete Genomes”的研究论文,构建了首个端粒到端粒的阿联酋人群二倍体泛基因组,成功解析了以往难以测序的基因组区域,并准确捕捉了阿联酋人群独特的遗传特征。

一、参考基因组组装的样本选择
阿联酋基因组参考由58个高质量组装构成,其中包括1个女性T2T组装和57个其他组装,形成首个阿联酋泛基因组数据集。样本来源于27个亲代-子代三人家系和30名无亲缘个体,经过精心筛选以充分代表阿联酋人群的遗传多样性:
(1)所有个体均选自按阿联酋人群祖先成分分层的不同种群簇;T2T参考样本被选用于代表主要祖先成分谱系中最高混合比例的个体;
(2)所有个体均无已知的先天性疾病,从而为基因组分析提供健康的基线;
(3)三人家系样本仅限于父母之间无亲缘关系的个体,其亲缘系数不高于四等亲,以确保遗传分型的准确性。
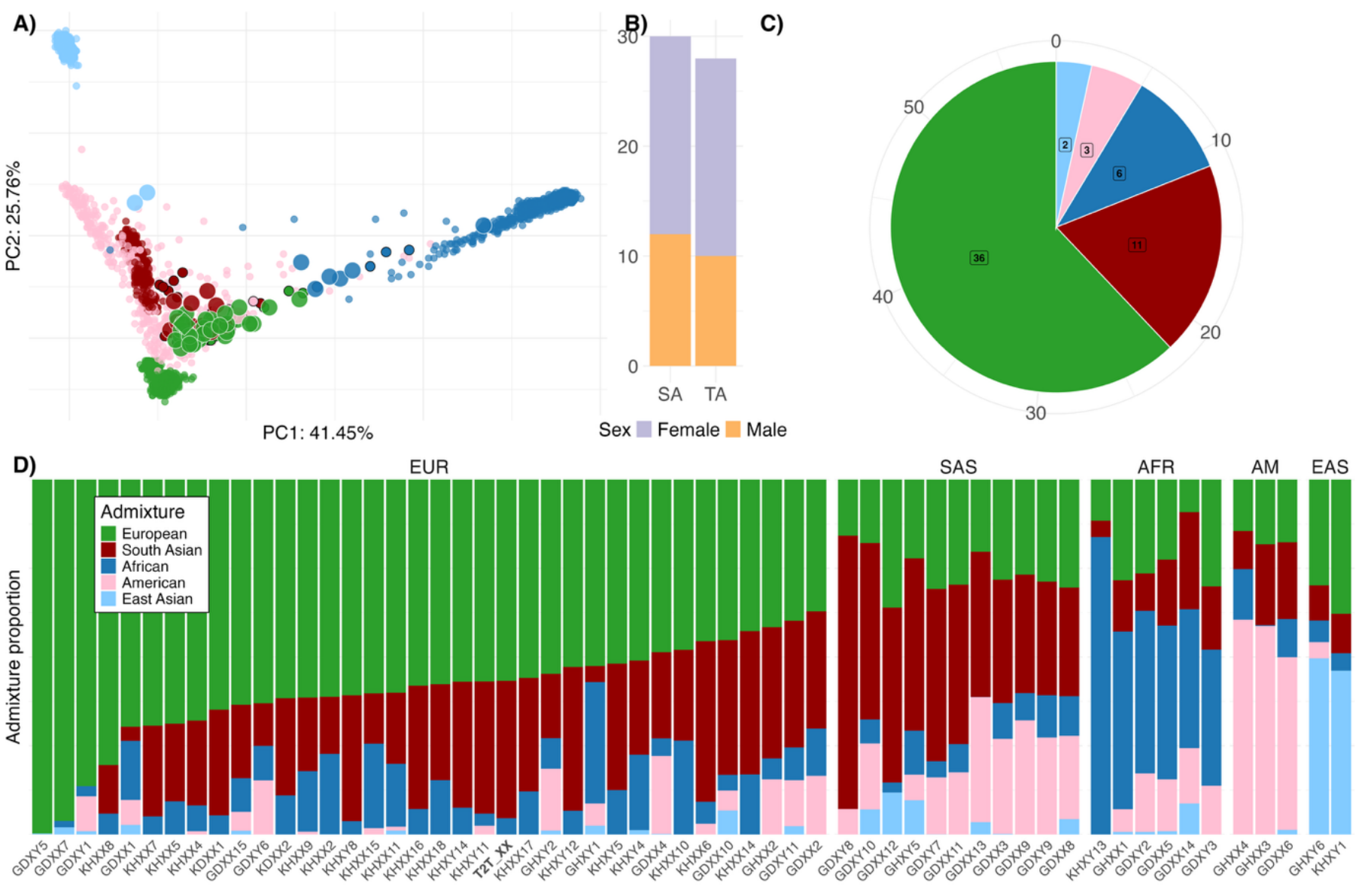
图1:阿联酋基因组参考中包含的58个基因组的基本特征和基于祖先的特征
二、端粒到端粒的组装
本研究构建了首个基于三人家系的阿联酋女性二倍体端粒到端粒基因组组装,命名为T2T-ERG_XX。组装整合了三种测序技术—Illumina、PacBio HiFi 和 ONT 超长测序,分别实现 147.75×、126.63× 和 111.8× 的覆盖度;母系样本与父系样本的 PacBio HiFi 覆盖度分别为 64.05× 和 63.01×。得到长度为3.03 Gb 的染色体级单倍型组装,来自母系与父系单倍型的QV值分别为 60.8 和 59.8,其中母系样本4、6、12、14、15、18、21、22号染色体及父系样本6、8号染色体实现了完整的T2T组装。整体组装覆盖CHM13 序列的94.4%,并完整包含 96.6% 的基因。在与 CHM13 比较后,发现少量着丝粒区域未能对齐,主要位于 13、14、15、18、21 和 22号染色体上。
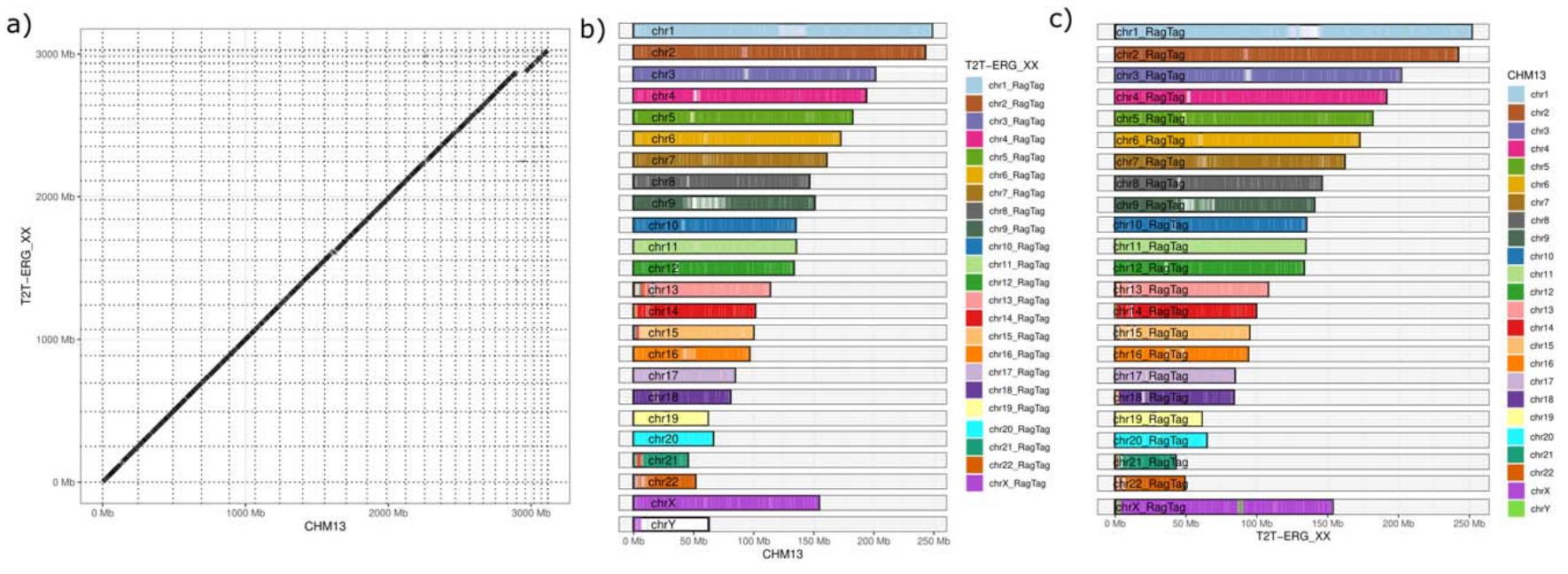
图2:T2T-ERG_XX母系单倍型与CHM13v2.0的比对
三、57个near-T2T二倍体组装
在已有阿联酋女性T2T基因组的基础上,作者进一步补充了57例具有代表性的个体样本,构建出高质量的阿联酋二倍体泛基因组,各样本平均测序深度为31.8×,N50中位读长为16.8kb。作者利用hifiasm结合三人家系数据生成完整的分型单倍型,并经ntLink、RagTag与Inspector等多阶段优化,最终获得染色体级组装。组装出的单倍型中位长度达3.02Gb,平均N50为150.32Mb,其连续性与CHM13基因组高度一致,误组装极少。比对至CHM13基因组后,样本中共检出其95.83%的基因,多拷贝基因检出率达76%。由于含有Y染色体,男性父系单倍型略短(2.93Gb),而女性单倍型保持在3.02Gb。Switch错误率中位数为0.33%,Hamming错误率为0.9%,均优于HPRC泛基因组水平。

图3:57 个后处理HiFi组装体的组装特征
四、阿联酋泛基因组变异分析
作者利用Minigraph-Cactus流程构建了两种泛基因组图,分别以CHM13v2和GRCh38为参考骨架,整合了T2T-ERG\_XX单倍型和114个阿联酋个体单倍型数据。结果显示,CHM13v2骨架图在各项指标上均显著优于GRCh38骨架图。这种优势在大型染色体和复杂区域(如13-22号染色体)尤为明显,例如13号染色体的节点数增加了28%。在变异分析方面,CHM13v2骨架图捕获了更多的遗传变异,特别是在单核苷酸变异和多等位基因位点的检测上表现更优。新增的58个阿联酋个体贡献了222.69 Mb新序列,其中566159bp为核心共享序列。覆盖度分析表明,CHM13v2骨架图中常染色体的平均覆盖度达到94.53%,其中21号染色体最高(99.4%)。性染色体的覆盖度差异显著:XX单倍型的chrX覆盖度为98.4%,而XY个体降至86.2%;chrY的覆盖度呈现碎片化特征。与传统比对方法相比,基于泛基因组图的SNV检测数量较少,且非洲血统个体显示出最高的SNV数量。
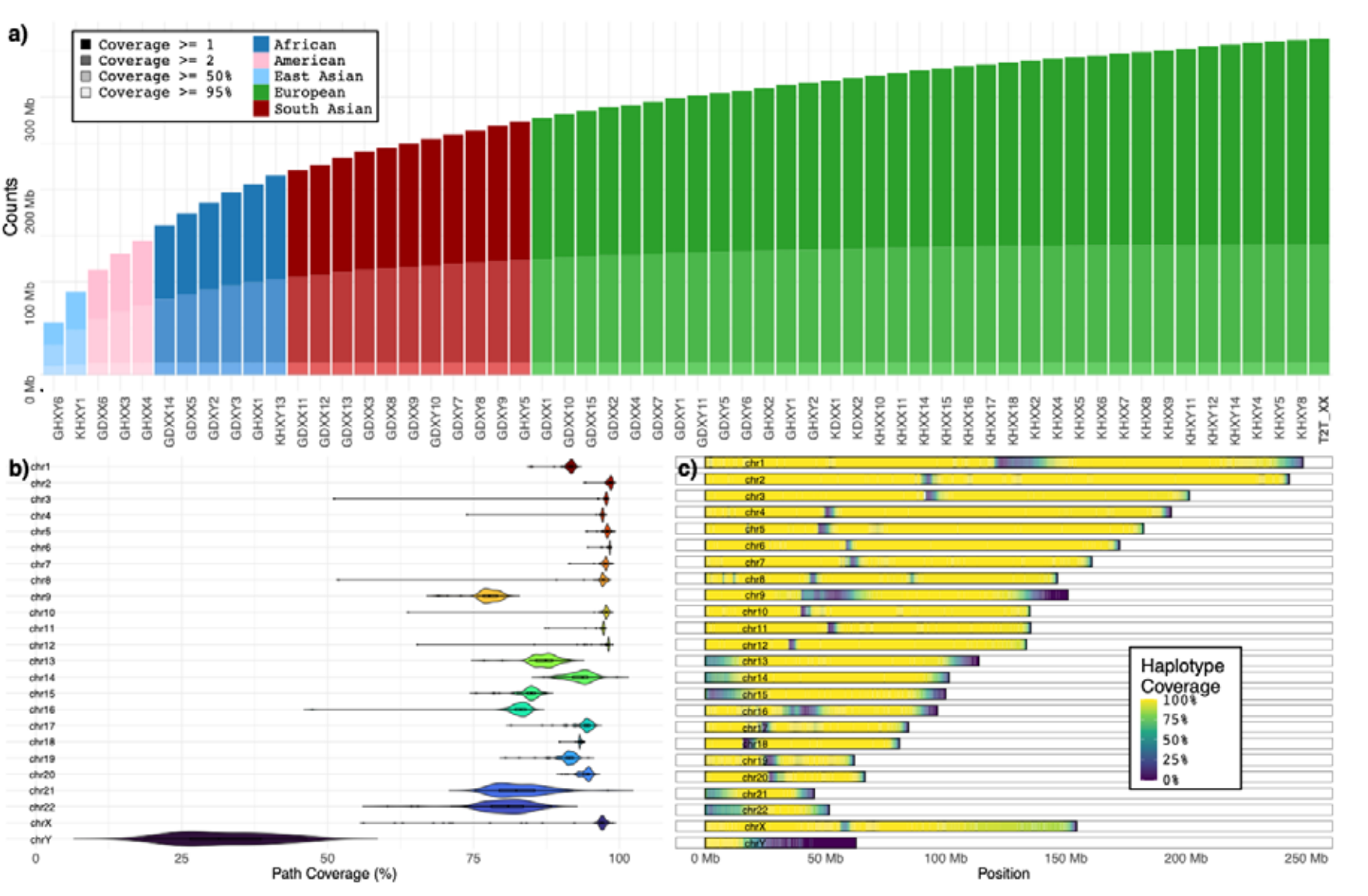
图4:泛基因组图谱特征
五、复杂 HLA 结构变异位点特异性分析
作者针对阿联酋泛基因组中HLA区域的临床相关复杂结构变异进行了特定位点分析,采用基于图的组装来解析大型多等位结构变异,并评估其相对于线性参考基因组(GRCh38、CHM13)的表现。结果表明,在阿联酋T2T泛基因组中存在五种环状结构(L1-L5),反映出跨越10个关键HLA关联基因的不同结构单倍型。L1是携带HLA-Y假基因的65.8kb插入,L4是包含HLA-H的44.0kb替代路径,这些大型环状结构的存在表明存在 GRCh38 或 CHM13v2 中未被代表的单倍型。L1的HLA-Y插入在16个单倍型中出现;跨HLA-H、HCG4B、HLA-K和HLA-U的多基因缺失在12个单倍型中检测到。L4单倍型(HLA-H插入)仅在一个个体中出现。按父母来源分析,母系单倍型对HLA-Y插入和多基因缺失的贡献均高于父系。总体来看,作者的发现与HPRC的结果一致但更具扩展意义:阿联酋人群中约65kb的HLA-Y插入频率较低,并且揭示了HPRC数据集中未捕获的结构复杂性,包括罕见的HLA-H纯合单倍型。
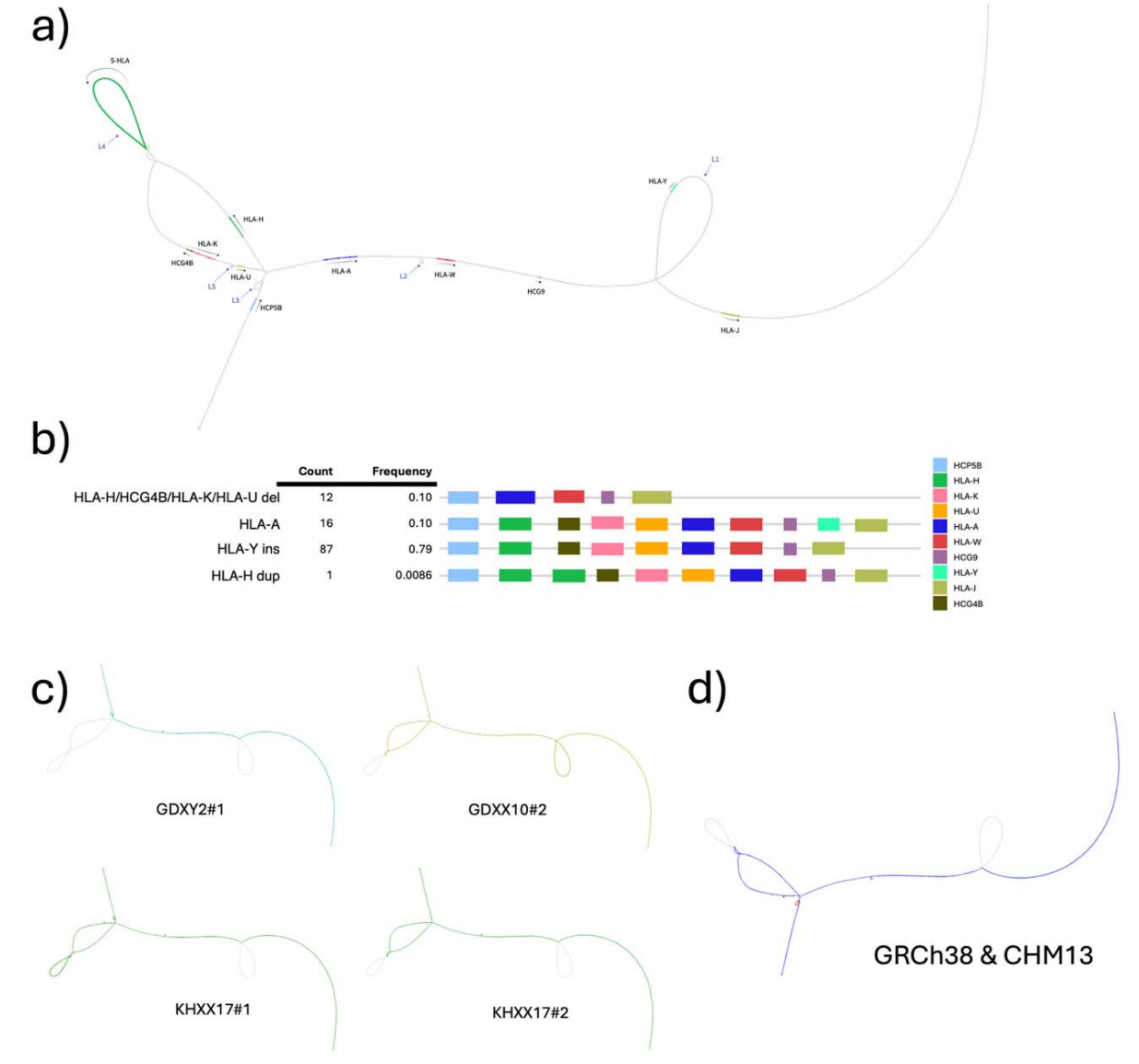
图5:可视化复杂泛基因组位点
六、基于阿联酋参考基因组的变异检测
作者选取了 119 份阿联酋个体基因组样本,对多种参考图谱的性能进行了系统评估。共比较五种配置:线性参考 GRCh38、T2T-CHM13、HPRC 泛基因组图,以及分别以 hg38 和 CHM13 为骨架构建的两种阿联酋群体特异性图谱。结果显示,GRCh38 的比对率最高(93.6% 的读取、94.7% 的碱基),CHM13 仅低约 0.1%。虽然三种基于图的参考平均少比对约 1% 的读取,但重复比对减少约 150 万条(约 2%),独特覆盖深度仍超过 30×。
相较于 GRCh38,基因组图参考可使单核苷酸变异(SNV)和小插入/缺失(indel)检出量分别增加约 1.9% 和 1.8%,且在非重复区域中也呈一致提升,表明变异检出灵敏度确有提高。使用 CHM13 参考时,变异检测性能已能达到图谱方法提升效果的一半,而将参考图谱特定化为阿联酋群体对变异数量的影响不足 0.2%,说明高质量的通用参考已能覆盖阿联酋人群的大部分短读变异多样性。在结构变异检测中,基于 GRCh38 的插入/缺失事件中位数约为 16.5 万个,而使用CHM13 或泛基因组图后减少约 96%,表明多数此类变异为比对伪影。总体来看,采用CHM13或图谱参考在比对率仅略降约1%的情况下,可提升约2%的小变异检测灵敏度,显著降低重复比对,并几乎完全消除伪结构变异。
七、HG001–HG007样本的基准分析
针对七个GIAB样本(HG001–HG007),研究分别利用短读与长读测序数据,在三种参考体系上进行了基准评估:线性GRCh38、HPRC图谱,以及以GRCh38为骨架的阿联酋T2T泛基因组图。结果显示,SNV检测在不同参考和平台间已趋于饱和,差异极小,说明从线性参考到图谱参考的转变对SNV调用精度影响有限。
相较之下,indel检测更依赖参考类型与读长。长读在GRCh38上的F1得分为0.9937,高于HPRC图谱(0.9803)和阿联酋图谱(0.9770),对应更多真阳性与更少假阳性。短读结果相近(0.9928–0.9931),图谱在轻微精度损失下恢复部分变异。总体而言,读长仍是主要影响因素,长读能显著提升召回率并减少假阴性。
结语
研究基于58个高质量二倍体组装(含1个女性T2T基因组),整合短读、长读及超长读测序,生成连续性与CHM13相当、误组装极低的高精度参考。团队进一步构建以CHM13与GRCh38为骨架的阿联酋泛基因组图,发现CHM13框架在结构复杂区域表现更优,可捕获更多真实变异并显著减少伪结构信号。特别是在HLA区,揭示了多种未被现有参考覆盖的复杂结构单倍型。在变异检测评估中,CHM13与图谱参考在比对率轻微下降的情况下,小变异灵敏度提升约2%,假阳性与重复比对显著减少,几乎消除了伪结构变异。与HPRC泛基因组相比,阿联酋特异图谱的性能差异不足0.3%,说明高质量的通用参考已能覆盖该人群主要变异特征。整体而言,该成果不仅填补了中东地区人群基因组空白,也为精准医学与人群遗传研究提供了新的高分辨率参考框架。
华命生物产品服务一览
华命生物目前已开通微信公众号、抖音、知乎、B站、小红书等线上平台,欢迎感兴趣的老师扫码关注了解更多内容!