NG重磅|玉米泛基因组解码抗旱基因
干旱是制约全球农业生产的主要环境胁迫之一,严重威胁作物产量与粮食安全。玉米对水分供应高度敏感,干旱胁迫显著降低其产量和稳定性。以往研究多利用GWAS鉴定抗旱相关基因,但仅依赖SNP信息,难以全面解析复杂性状的遗传基础。结构变异(SV)在基因功能调控与表型形成中发挥关键作用,然而由于玉米基因组高度复杂(重复序列约占85%)且遗传多样性极高,基于单一参考基因组的SV检测往往存在局限,难以准确捕获群体层面的结构变异信息。
中国农业大学秦峰研究团队于2025年10月27日在国际著名期刊Nature Genetics上发表了一篇题目为“A pangenome of maize provides genetic insights into drought resistance”的研究论文,整合了来自56份玉米种质的基因组数据,构建目前最全面的玉米泛基因组,并系统解析了玉米抗旱性的遗传基础。

一、玉米抗旱性的遗传多样性
作者首先系统评估了25个来自全球不同生态区的玉米种质的抗旱遗传多样性。通过幼苗存活率(SR)和开花期花丝-雄穗间隔(ASI)及籽粒产量等指标,分析了种质在干旱胁迫下的抗旱表现。结果显示,约36%的种质具较强抗旱性,30%为中度抗旱,34%为易感型,其中CIMBL55表现最耐旱,Mo18W最敏感。ASI与产量呈显著负相关,而SR与产量无明显关系。包括GEM55、CIMBL55等在内的部分种质在不同生育阶段均表现出稳定的抗旱性和较高产量,被鉴定为抗旱型种质。
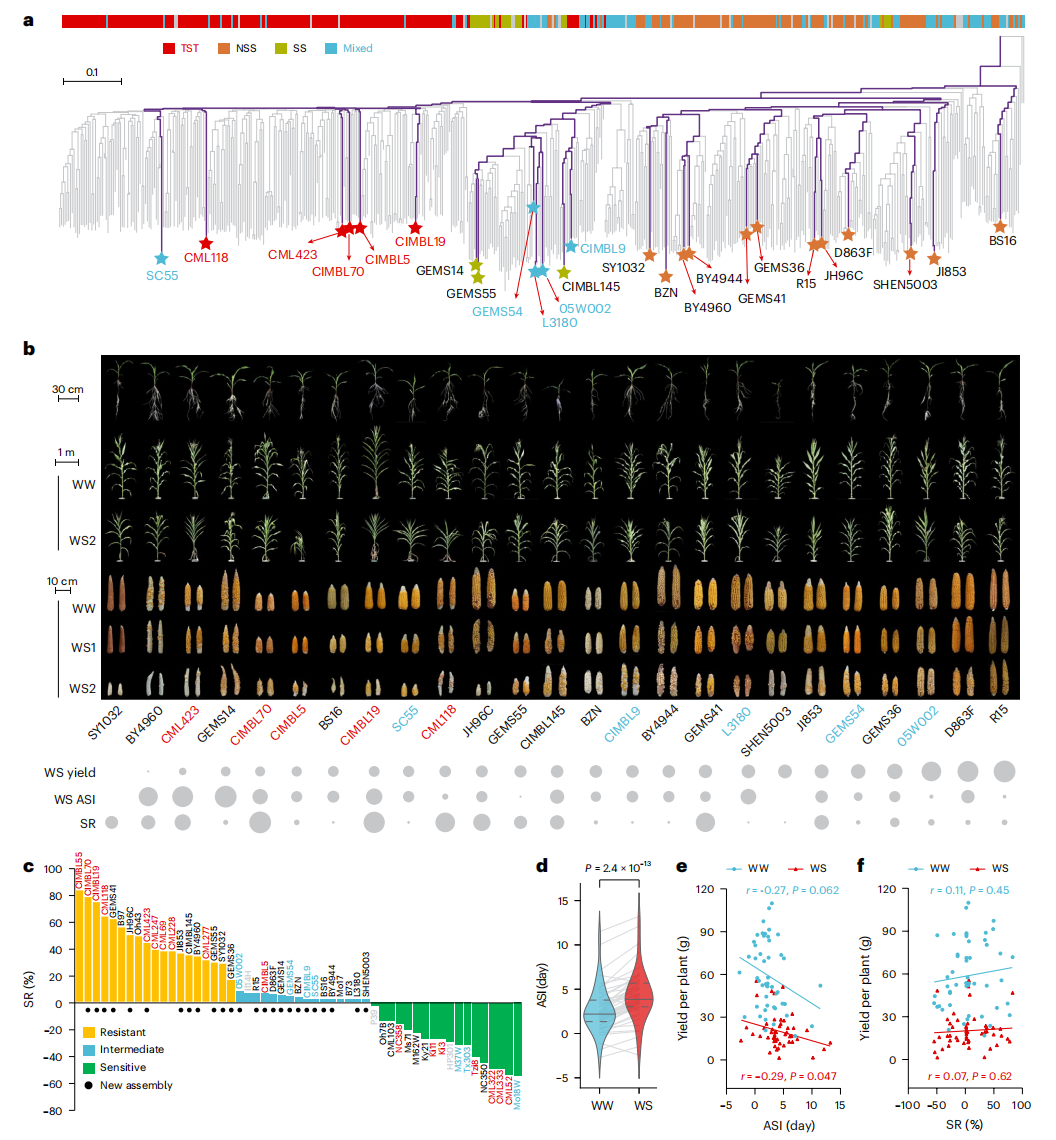
图1:25份玉米种质资源的系统发育分析及其干旱相关表型特征
二、25个玉米基因组的从头组装和注释
本研究基于Oxford Nanopore(98×)和Illumina(60×)测序技术,对25个玉米种质进行了从头基因组组装与功能注释。结果显示,组装的contig N50平均为71.04 Mb,基因组大小约2.17 Gb,97.7%的序列成功锚定到染色体。BUSCO分析表明,平均96.49%的植物基因完整组装,基因注释完整性达96.98%,显示出高质量的组装水平。平均注释到39880个蛋白编码基因,LTR组装指数超过20,表明组装连续性极高。基因组中85%以上为重复序列,其中主要为反转录转座子。
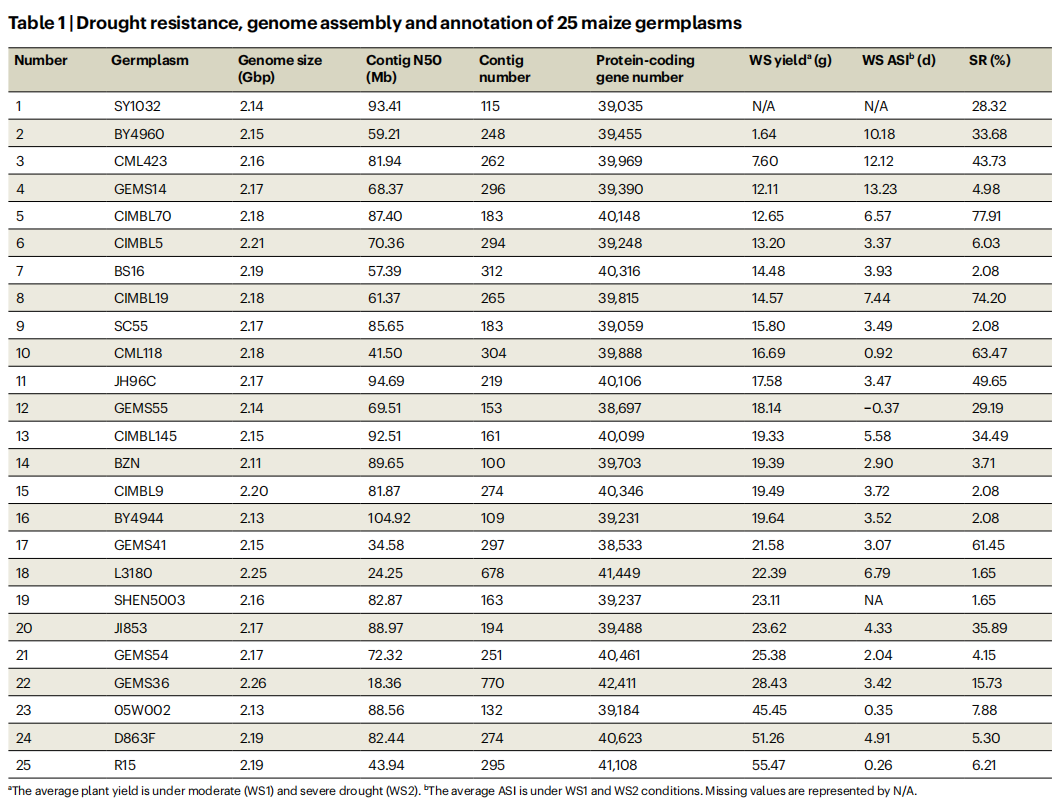
表1:25份玉米种质的抗旱性、基因组组装与注释
三、ABA相关或干旱相关基因的泛基因组视角
研究整合56份玉米种质,共识别出126384个泛基因,其中核心与亚核心基因家族占37.9%。研究重点分析了1344个ABA(脱落酸)或干旱相关基因,包括信号感知(PYLs)、转导(SnRK、PP2C、RBOH)及转录调控(WRKY、MYB、NAC等)等基因。结果表明,这些基因中超过92.4%为核心或亚核心基因,显示其在进化中的高度保守性。通过多算法联合比对,研究共鉴定出约297万个非冗余SVs,主要分布于基因间区,其中部分稀有变异(如ZmNCED5串联重复、ZmDRO1缺失、ZmNAC111提前终止突变)与抗旱性状密切相关。
研究在368份玉米种质中对SV进行基因分型,构建了两套群体SV集(pSV-1和pSV-2)。通过GWAS与eQTL进一步分析发现,52.5%的ABA或干旱相关基因的表达受遗传变异显著影响。利用pSV-2数据估算多种性状(代谢物、基因表达、幼苗形态)在干旱与正常条件下的遗传力,结果显示SV数据比SNP或小型插缺能更好解释复杂性状的遗传基础。
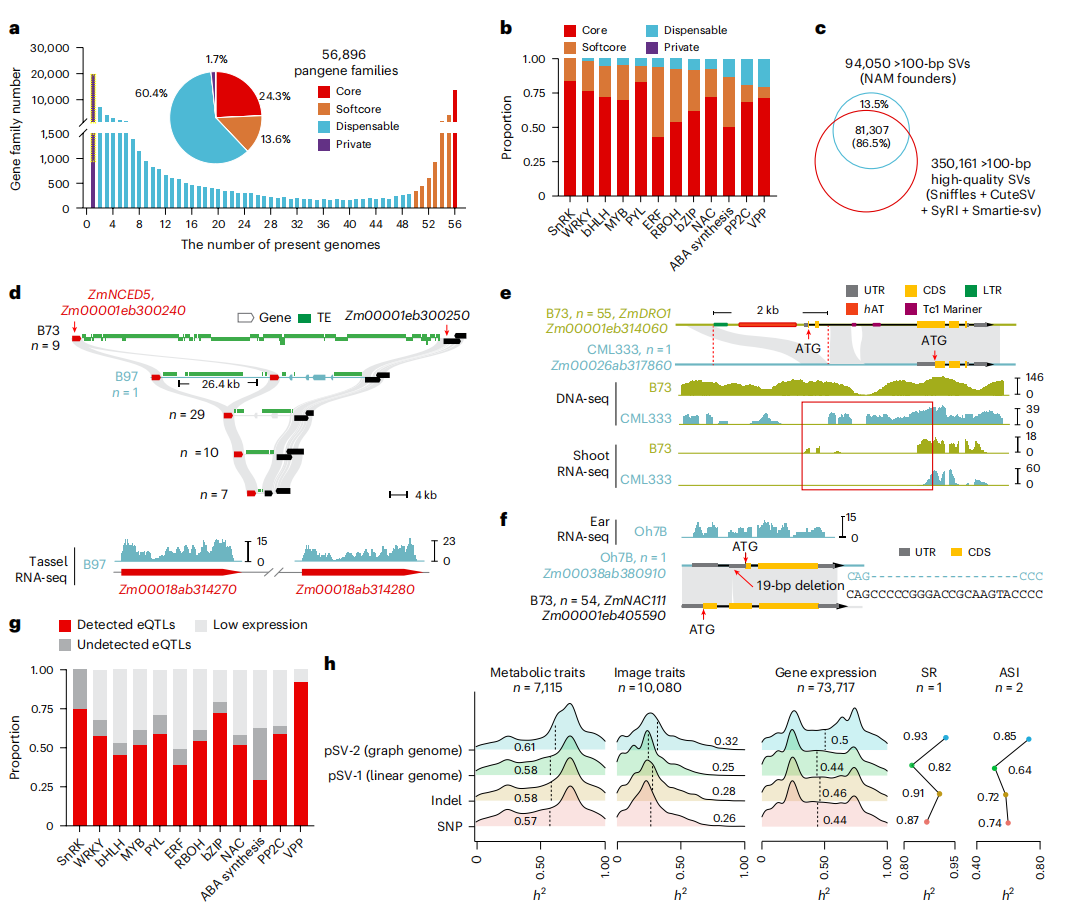
图2:玉米泛基因组分析及aba相关和干旱相关基因变异
四、ZmUGE2与ZmSIL2在幼苗期抗旱中的作用
作者基于对1344个ABA/抗旱相关基因进行eGWAS(表达量关联分析),利用群体水平可分型的SV与基因表达差异建立关联,从而筛选出与抗旱性密切相关的候选基因ZmUGE2和ZmSIL2进行功能验证。
研究发现,在干旱胁迫下ZmUGE2的多个变异位点与基因表达水平及存活率显著相关,携带Hap2型的种质在干旱后表现出更高的表达量和更强的抗旱性,与抗旱性呈现正相关。ZmUGE2编码UDP-葡萄糖4-差向异构酶,参与细胞壁的糖组分转化。过表达植株(ZmUGE2–OE)细胞壁更厚、抗旱性更强;而敲除植株(zmuge2–KO)细胞壁变薄、抗旱性显著下降。干旱会短暂诱导该基因表达并促使蛋白积累,作者认为其可能通过增强细胞壁机械强度、维持细胞完整性来抵御胁迫。
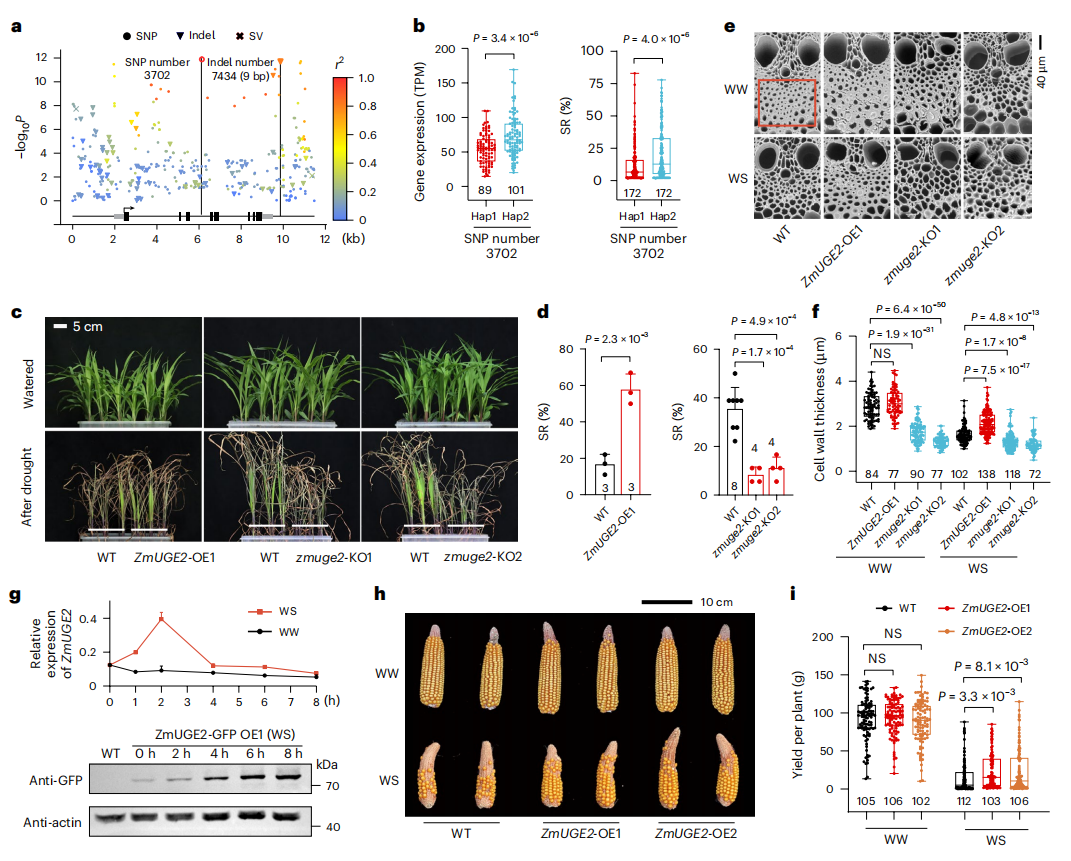
图3:ZmUGE2正调控抗旱性
另一基因ZmSIL2编码三螺旋转录因子,与拟南芥ASIL2高度同源。进一步分析发现其负向调控抗旱性:表达量低的Hap2型种质反而在干旱下存活率更高。在其3′-非翻译区检测到多种结构变异,形成多个单倍型亚群,且这些变异在玉米祖先类群中已存在,表明该基因在驯化前已发生分化。功能验证显示,ZmSIL2–OE植株抗旱性降低,而CRISPR敲除株(zmsil2–KO)抗旱性增强。转录组与ChIP-seq分析鉴定出292个其直接调控的干旱应答基因,识别基序为CGCCGCCC。其中包括ZmAZF2与AP2/EREBP等应激相关转录因子,说明ZmSIL2通过调控应激基因网络来影响植株的抗旱性。
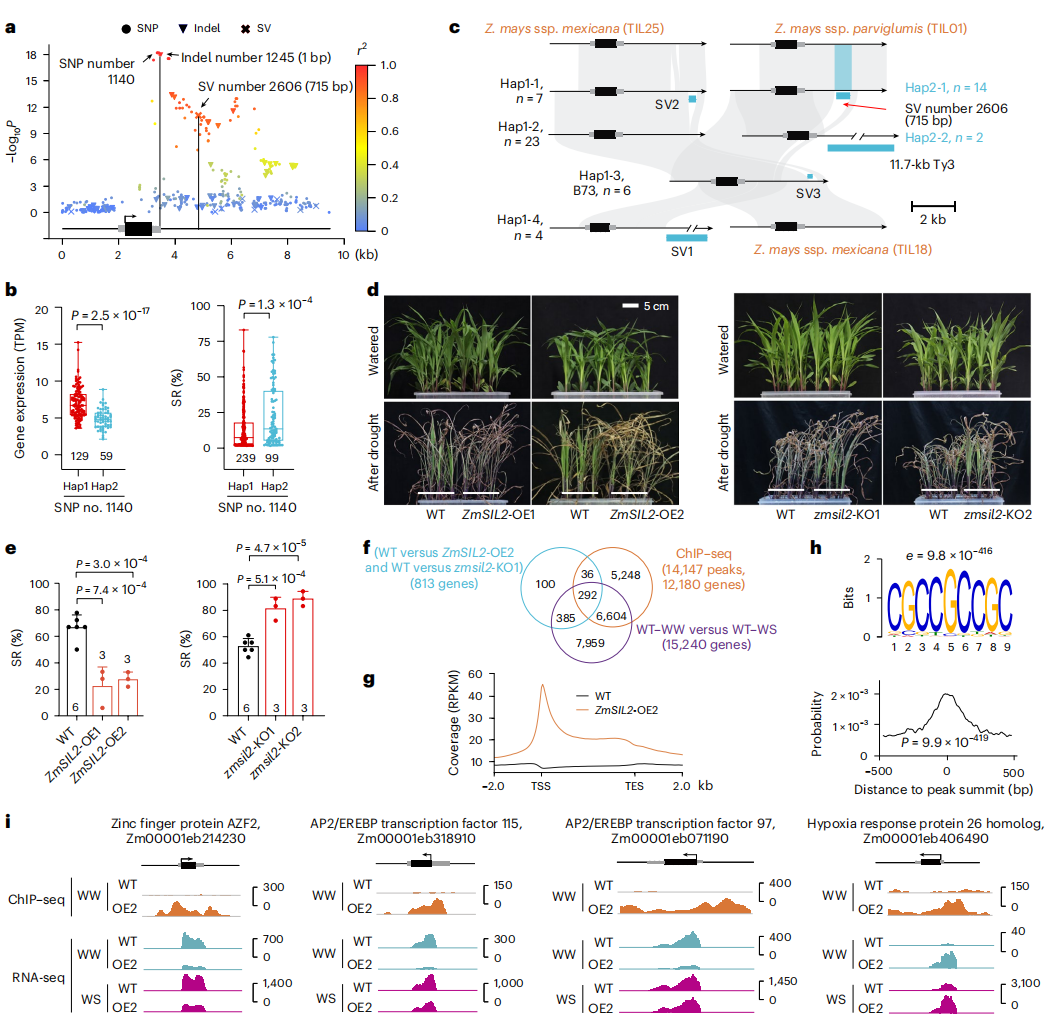
图4:ZmSIL2在苗期负调控抗旱性
五、ZmASI3 在玉米开花期抗旱中的作用
在开花期抗旱研究中,作者通过GWAS分析鉴定出位于染色体3(150.63-150.67 Mb)的ZmASI3基因对花丝抽出间期(ASI)的变异具有显著影响。该基因编码含SNF2解旋酶结构域的蛋白,可能参与染色质重塑。作者基于以上结果将种质划分为HapA和HapC两类,发现HapA型在干旱和正常条件下ASI均较短,抗旱性更强。ZmASI3可产生两种转录异构体:T01能正常剪接形成全长蛋白,而T02因内含子保留导致翻译提前终止并生成无功能截断蛋白。生化与细胞实验验证发现,仅全长ZmASI3具备ATP酶活性与染色质重塑能力。泛基因组分析揭示,该内含子中存在多种结构变异,可能影响剪接效率。CRISPR–Cas9敲除实验进一步验证,zmasi3-KO植株在干旱条件下ASI显著延长且产量降低。综上,作者认为ZmASI3通过介导染色质重塑正向调控玉米开花期抗旱性,而其内含子变异可能削弱该功能。
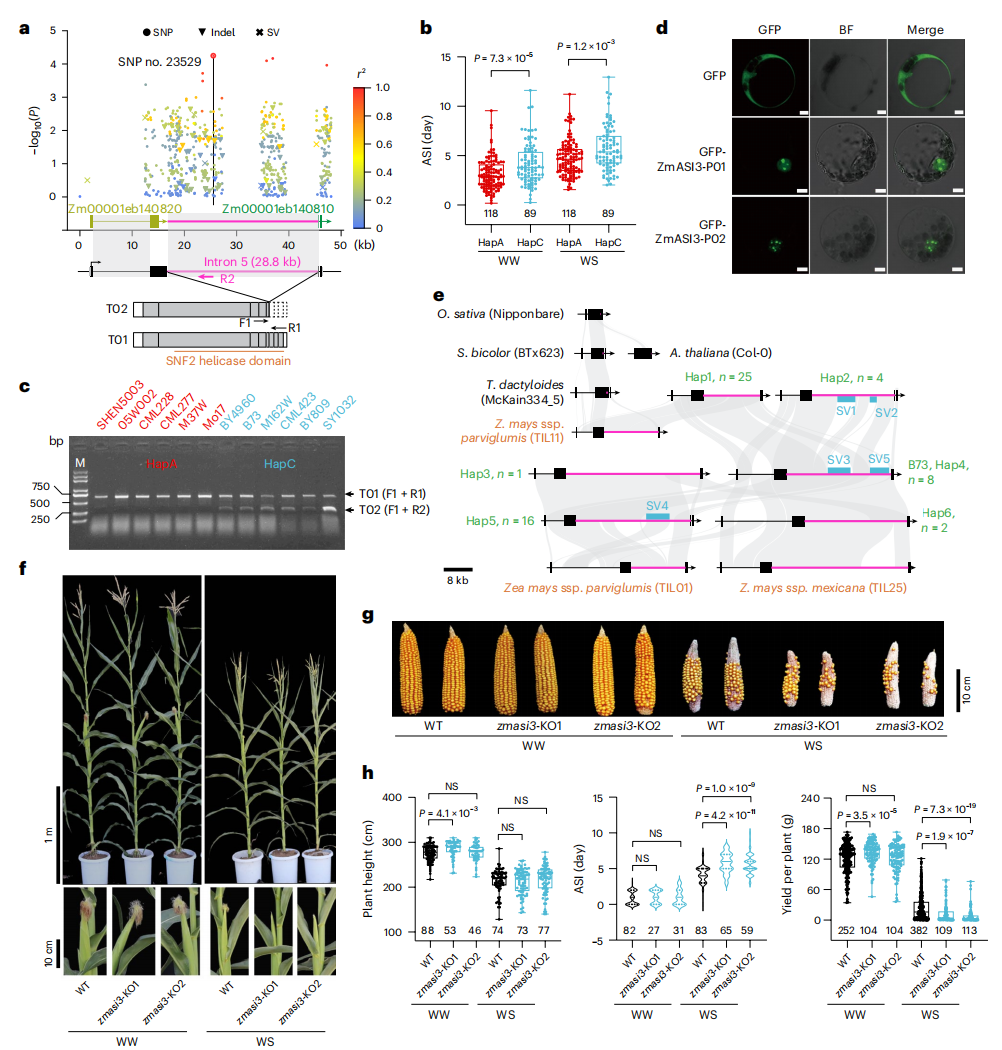
图5:ZmASI3基因有助于花期抗旱性的提高
结语
本研究从头组装25份玉米种质基因组,并与26个NAM创始系及5份已发表基因组联合评估抗旱性表现,构建含56个基因组的玉米泛基因组资源,系统解析了玉米抗旱性的遗传基础。作者发现抗旱相关基因大多为核心基因,抗旱差异主要源于调控变异而非基因缺失。结合GWAS与eQTL分析,鉴定出三种关键基因:ZmUGE2增强细胞壁沉积、维持细胞结构稳定,从而提升抗旱性;ZmSIL2为负调控因子,通过抑制AP2/ERF类转录因子表达降低抗旱能力;ZmASI3在开花期抗旱中发挥正向作用,其大型内含子结构变异影响剪接效率并削弱功能。CRISPR验证表明,三基因在干旱条件下显著影响植株生长与产量。研究在泛基因组框架下揭示了玉米抗旱的分子机制,提供了精准育种的新靶点与理论基础。
华命生物产品服务一览
华命生物目前已开通微信公众号、抖音、知乎、B站、小红书等线上平台,欢迎感兴趣的老师扫码关注了解更多内容!