NC重磅|泛基因组解析茄子关键性状与演化轨迹
茄子 (Solanum melongena) 是全球重要的茄科蔬菜作物,年产量超过5900万吨,中国和印度为主要生产国。与多起源于新大陆的茄科作物不同,茄子起源于亚洲,在系统发育上与番茄、马铃薯等作物存在较大差异。栽培种S.melongena与其直接野生祖先S.insanum及近缘种S.incanum构成明确的演化谱系,蕴含丰富的遗传多样性。
国外研究团队于2025年11月11日在Nature Communications上发表了题为“Graph-based pangenomes and pan-phenome provide a cornerstone for eggplant biology and breeding”的研究论文,构建了覆盖栽培与野生茄子遗传多样性的核心材料库,并基于长短读测序建立图谱化泛基因组;同时通过泛基因组关联分析系统解析了茄子的演化历史及刺、抗病性和代谢物含量等关键性状的遗传机制。

一、全球茄子核心种质库的构建与基因组组装
研究基于来自105个国家的3412份全球茄子材料,构建了包含368份代表性材料的全球茄子核心种质库,其中涵盖栽培茄子与其野生近缘种S.insanum和S.incanum。研究团队对全部材料开展短读长测序,并对40份具有代表性的材料进行了Nanopore长读长测序和部分Hi-C测序,获得高质量染色体级基因组,基因组大小约1.10-1.14Gb,BUSCO完整度达98%左右,均达到参考级别。基因组分析表明,重复序列占比超过70%,以LTR-RT为主。系统发育分析显示,栽培种与两种野生种之间的关系受到杂交、基因渗入等因素影响,其分化时间约为0.75Mya与0.29Mya。
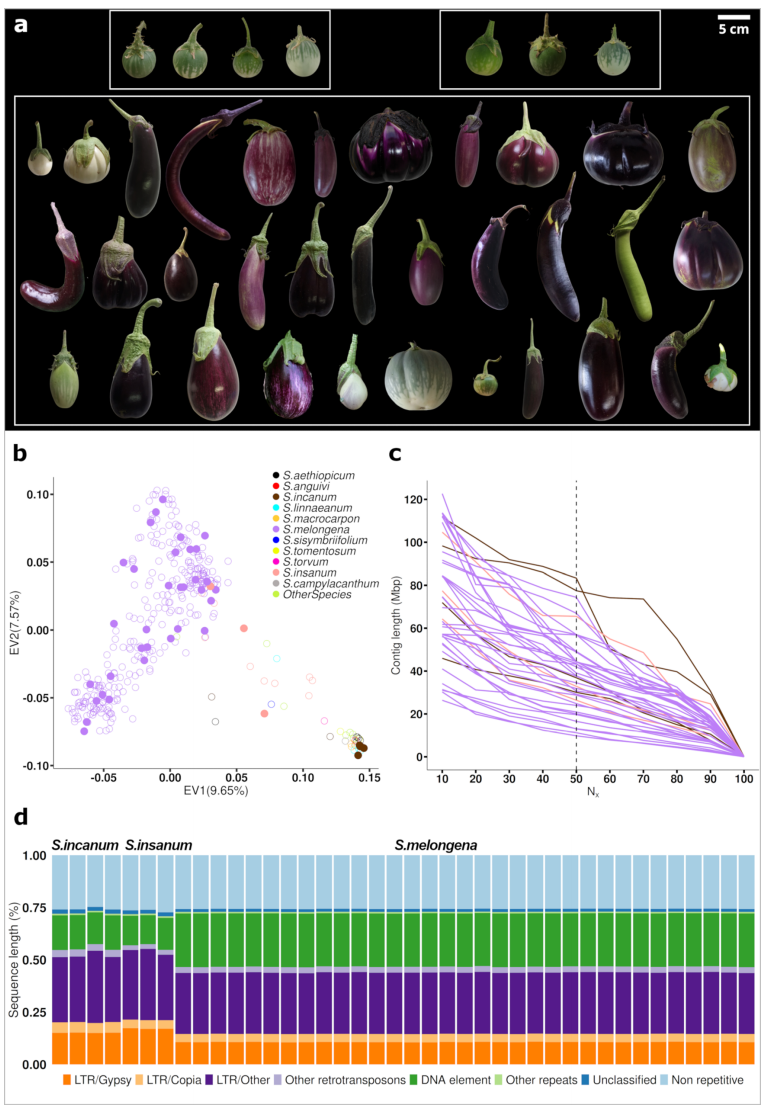
图1:40个茄子参考基因组
二、系统发育与共线性分析
基因家族演化分析显示,不同物种在关键生物学功能上呈现出明显而具方向性的差异:S.melongena的基因扩张主要集中在胁迫适应相关功能,如基因沉默、脱水耐受及角质层蜡合成;而S.insanum则在解毒与萜类化合物合成途径上出现显著扩张,同时伴随部分生长相关通路的收缩;S.incanum更偏向于生长与生殖过程的基因扩张,但在乙烯代谢与病害响应相关基因上呈收缩趋势。在共线性与基因重复层面,野生种普遍表现出更频繁的结构变异和更丰富的发育相关基因重复,体现其较高的基因组可塑性与生态适应能力。而与此相比,栽培茄子在能量代谢及脱落酸(ABA)响应等胁迫相关路径中呈现独特的基因富集模式,说明其驯化过程可能强化了应对栽培环境压力的能力。
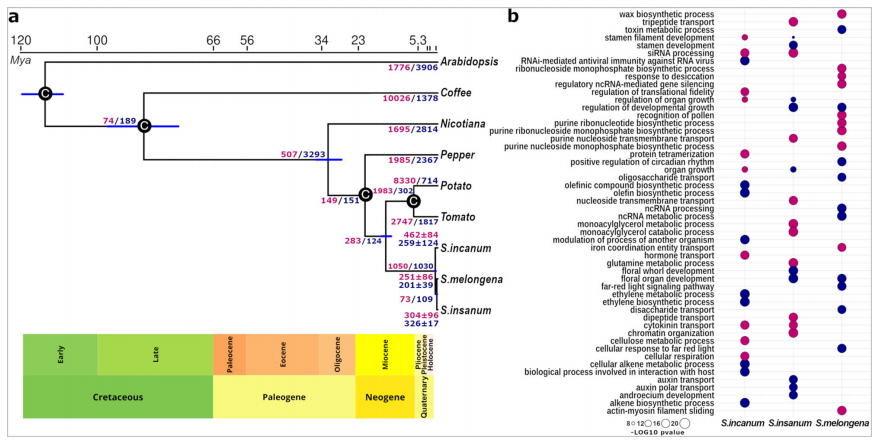
图2:茄子及其近缘种分化时间及基因家族演化

图3:40 份茄子参考基因组的同线性与基因重复分析
三、基于图谱的茄子泛基因组
为全面揭示茄子类群的遗传多样性并避免对单一参考基因组的偏倚,基于40份染色体级高质量基因组,构建了规模达2.94Gb的与参考无关的泛基因组(PG-SMA),较已有参考泛基因组扩大一倍以上,检测到8.3万余个结构变异。系统分析显示S.incanum与S.insanum并未形成独立类群,而是相互交错,与基因树结果一致。PG-SMA结构揭示约40%的可丢弃基因组与约50%的核心基因组,表明茄子类群基因组具有高度可塑性。
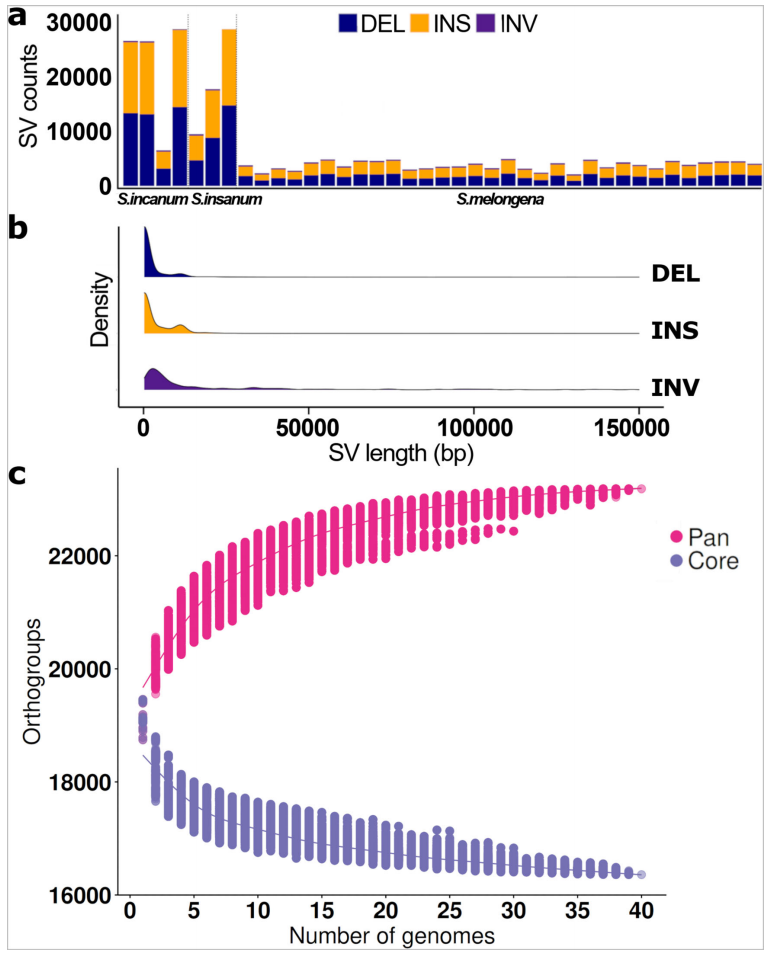
图4:茄子及其近缘种的全基因组图谱
四、栽培茄子及其近缘种的种群结构
对核心种质库的群体遗传分析共鉴定1289万个SNP和1.26万个结构变异。基于SNP的最大似然树,材料分为栽培类群S.melongena与以S.insanum、S.incanum为主的野生类群两大分支,部分S.insanum聚入栽培类群分支,说明可能存在历史基因交流。分析还识别出“Eggplant”“Anguivi”“Lasiocarpa”三大稳定亚群。SNP-PCA显示栽培类群聚集紧密,S.insanum分布较为分散并部分靠近栽培类群,S.incanum与部分野生种存在遗传重叠。
地理成分分析将栽培茄子材料分成“东南亚”和“印度次大陆”两大类,它们分别与各自区域的野生祖先S.insanum最为接近。这说明栽培茄子并非在单一地区起源,而是在这两个区域分别由当地的野生种独立完成了驯化。进一步的群体结构分析显示,S.insanum在不同地区呈现明显的遗传分化,马达加斯加材料则表现为来自亚洲的混合类型。东南亚栽培种中仍可检测到本地S.insanum的遗传成分,而印度栽培种则几乎缺乏此信号。野生类群整体保留独特基因背景,是茄子多样性利用的重要资源。

图5:茄子及其野生近缘种的系统发育与群体结构
五、种群动态与种群历史推断
作者结合SNP和SV数据,采用FST评估了不同种群间的基因分化。研究发现,茄子与其野生亲缘种S. insanum的基因分化程度最大,其次是与S. incanum的基因分化。在茄子种群中,来自东南亚、印度和日本的种质与S. insanum的基因亲缘关系最为密切,进一步支持东南亚作为茄子的主要驯化中心的推论。D统计分析进一步确认了从S. insanum向东南亚、印度和日本种质的非对称基因流动,而欧洲品种的基因内流较少。研究还分析了茄子的驯化与迁移历史,结果表明最可能的迁移路径为从S. insanum向东南亚茄子种质的基因流动,以及日本/韩国种质回流至S. insanum。人口学建模显示,S. insanum和S. incanum的有效种群数量大约在40-45千年前的冰川最大期经历了瓶颈,而茄子来自东南亚和印度的种质则在约15千年前遭遇了更为近期的瓶颈,符合茄子驯化的时间框架。
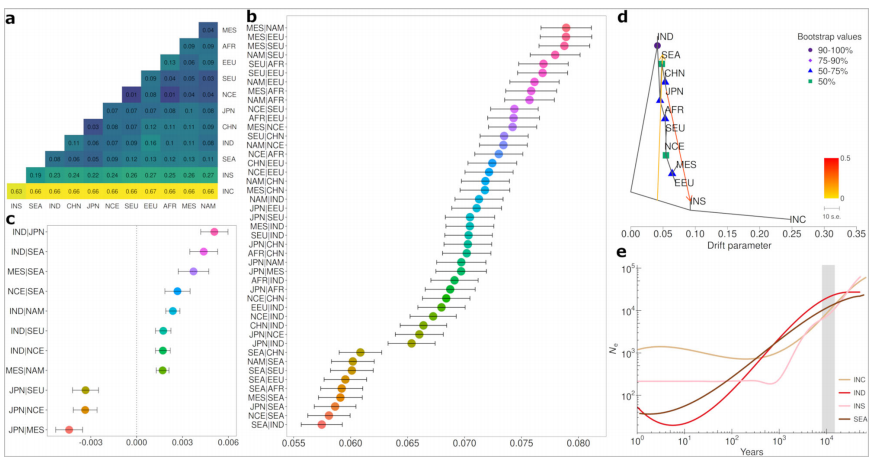
图6:基于PG-SMA SNPs的茄子驯化来源于S.insanum
六、茄子泛表型研究
研究通过在土耳其、意大利和西班牙三地对茄子核心种质进行随机区组试验,系统获取了农艺、生物/非生物胁迫及果实代谢等共218项表型数据。作者从中选取了具有代表性的性状,包括果萼和叶片刺毛(农艺变异)、对枯萎病的抗性(关键生物胁迫性状)以及果皮和果肉中绿原酸与异绿原酸含量(代谢特征)。为实现对321份材料的准确比对、多态性检测和泛基因组关联分析(Pan-GWA),研究团队基于33份茄子材料和参考基因组GPE001970构建了第二个泛基因组图谱 PG-SM,并与原图谱共同用于后续Pan-GWA 分析。
七、茄子泛gwa分析阐明了茄子多刺的遗传调控机制
作者基于该图谱,明确了6号染色体上的LOG3基因是控制刺毛形成的关键遗传因子。绝大多数材料中,“A”等位基因与有刺表型相关,而位于剪接位点的“G”等位基因则导致无刺表型,并伴随LOG3表达量下降。此外,研究还发现一个引起LOG3外显子缺失的226bp片段缺失,以及在野生材料中存在的涉及LOG3区域的染色体大片段易位,进一步揭示了无刺表型形成的复杂遗传机制。

图7:影响茄子叶片和花萼刺毛的突变
八、茄子抗镰刀菌的分子基础
为了探究茄子抗尖孢镰刀菌的关键分子机制,作者通过全基因组关联分析结合泛基因组结构变异分析在染色体2和11上鉴定到两个主要QTL,其中染色体11的RPP13-like抗病基因簇是核心抗性来源;染色体2的QTL对应一个260 bp插入,其邻近的AHL家族基因SMEL5_02g002450可能参与胁迫响应。泛基因组分析显示,易感材料中在SMEL5_11g023850第一外显子出现特异插入,并伴随约31 kb的大片段缺失,影响抗真菌蛋白基因SMEL5_11g023860及RPP13-like基因SMEL5_11g023870的完整性与表达。该缺失与感病高度相关,并在S. insanum与S. incanum中普遍存在,表明其为古老的结构变异并受到选择清扫作用。进一步分析显示,染色体2的插入在大多数野生近缘种中固定,而缺失该插入的材料通常易感;但只要染色体11上的RPP13-like基因簇保持完整,即便没有染色体2的插入依然可表现完全抗性。总体表明,茄子抗尖孢镰刀菌主要由染色体11的RPP13-like基因簇主导,并受到特定古老结构变异的调控。

图8:解读茄子的镰刀菌枯萎病抗性
九、茄子中绿原酸代谢的分子基础
研究进一步探讨了茄子中绿原酸及其衍生物的代谢机制,通过全基因组关联分析识别了8个QTL,最强的位于染色体6,但未发现明确的候选基因。研究表明环境因素,如温度和光照,可能影响绿原酸的合成。此外,研究在染色体4(4.88-5.55Mb)上鉴定了一个主要QTL,显著影响茄子果皮和果肉中的异绿原酸含量。该区域内发现了SMEL5_04g005040基因,携带突变导致蛋白质非功能化。分析显示,高异绿原酸含量的茄子种质携带一个单倍型,而低含量的种质则携带另一种。大多数野生亲缘种(如S.insanum、S.incanum)天然积累较高水平的异绿原酸,并携带高积累单倍型,只有S.incanumGPE008640携带低积累单倍型。作者认为SMEL5_04g005040是决定茄子及其野生亲缘种异绿原酸含量的关键基因。
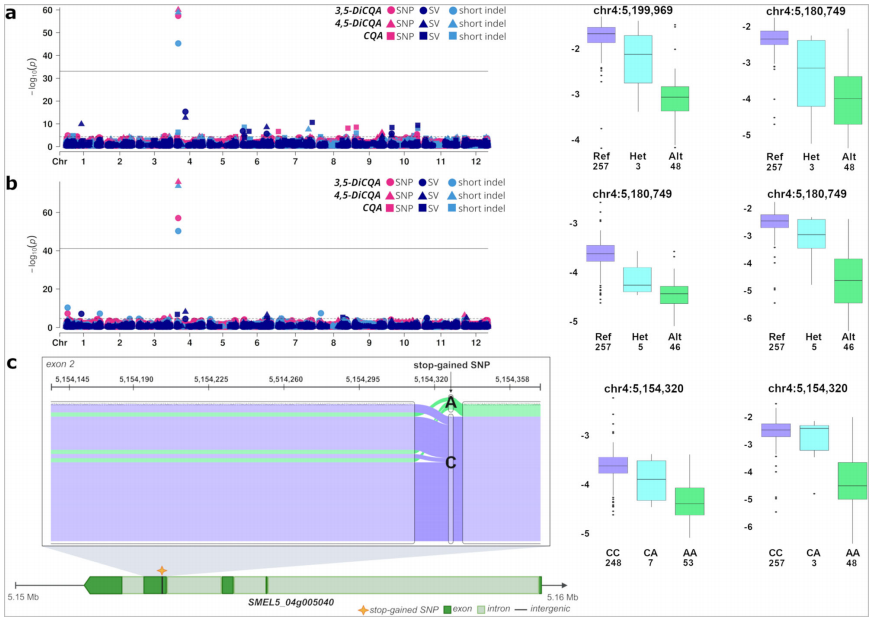
图9:绿原酸及其衍生物的泛gwa结果
结语
本研究通过构建茄子栽培种S. melongena及其野生亲缘种S. insanum 和 S. incanum的泛基因组,深入分析了茄子的遗传多样性和重要农艺性状的遗传基础。研究结合多个不同性状样本的短读和长读测序技术,构建了两个泛基因组图谱,并通过泛基因组关联分析(Pan-GWA)找到了多个关键性状的控制基因:6号染色体上的LOG3基因与茄子果实刺毛发育相关,11号染色体的RPP13-like抗病基因簇与茄子的抗枯萎病性状密切相关, GDSL脂肪酶样基因参与绿原酸代谢过程。研究还揭示了茄子在东南亚和印度次大陆的独立驯化过程,并且指出野生种与栽培种之间的基因流动对当前茄子遗传结构的形成起到了重要作用,为茄子的基因组学研究和未来基因组辅助育种提供了宝贵的遗传资源。
华命生物产品服务一览
华命生物目前已开通微信公众号、抖音、知乎、B站、小红书等线上平台,欢迎感兴趣的老师扫码关注了解更多内容!