Sci Data项目文章 | 阿纳鲳鲹单倍型T2T基因组
阿纳鲳鲹(Trachinotus anak)是我国重要的海水养殖经济鱼类,主要分布于西太平洋热带和亚热带海域。该物种具有肉质细嫩、生长迅速和环境适应性强等优点,近年来在我国东南沿海地区养殖规模迅速扩大,年产量居海水养殖鱼类前列。尽管已有阿纳鲳鲹基因组组装报道,但现有版本仍存在缺口多、端粒序列不完整等问题,限制了分子育种和功能基因组学研究的深入开展。
广东海洋大学水产学院陈华谱研究团队近日在Scientific Data上发表标题为“Telomere-to-telomere haplotype-resolved genome assembly of a female oyster pompano (Trachinotus anak)”的研究性论文,成功构建了雌性阿纳鲳鲹的T2T级单倍型分型基因组,其质量在连续性、准确性和完整性方面均显著优于此前发布的基因组版本。华命生物高级生信工程师陈容作为文章共同作者参与研究并承担了生物信息学分析相关工作。

一、基因组测序与组装
本研究构建了阿纳鲳鲹的两套高质量单倍型分辨染色体级参考基因组。研究共获得多类型测序数据,包括76.96 Gb的DNBSEQ-T7短读长数据、137.54Gb的PacBio HiFi长读长数据、85.72 Gb的Nanopore超长读长数据,以及 158.46 Gb 的 Hi-C 数据。基于k-mer分析,估计基因组大小为约641-642Mb,整体杂合度较低。最终获得的两套单倍型基因组分别为663.78 Mb(hapA)和661.09 Mb(hapB),均成功锚定至24条染色体。所有染色体均实现端粒到端粒连续性,未检测到组装缺口,表明该基因组在完整性和连续性方面均达到了较高水平。
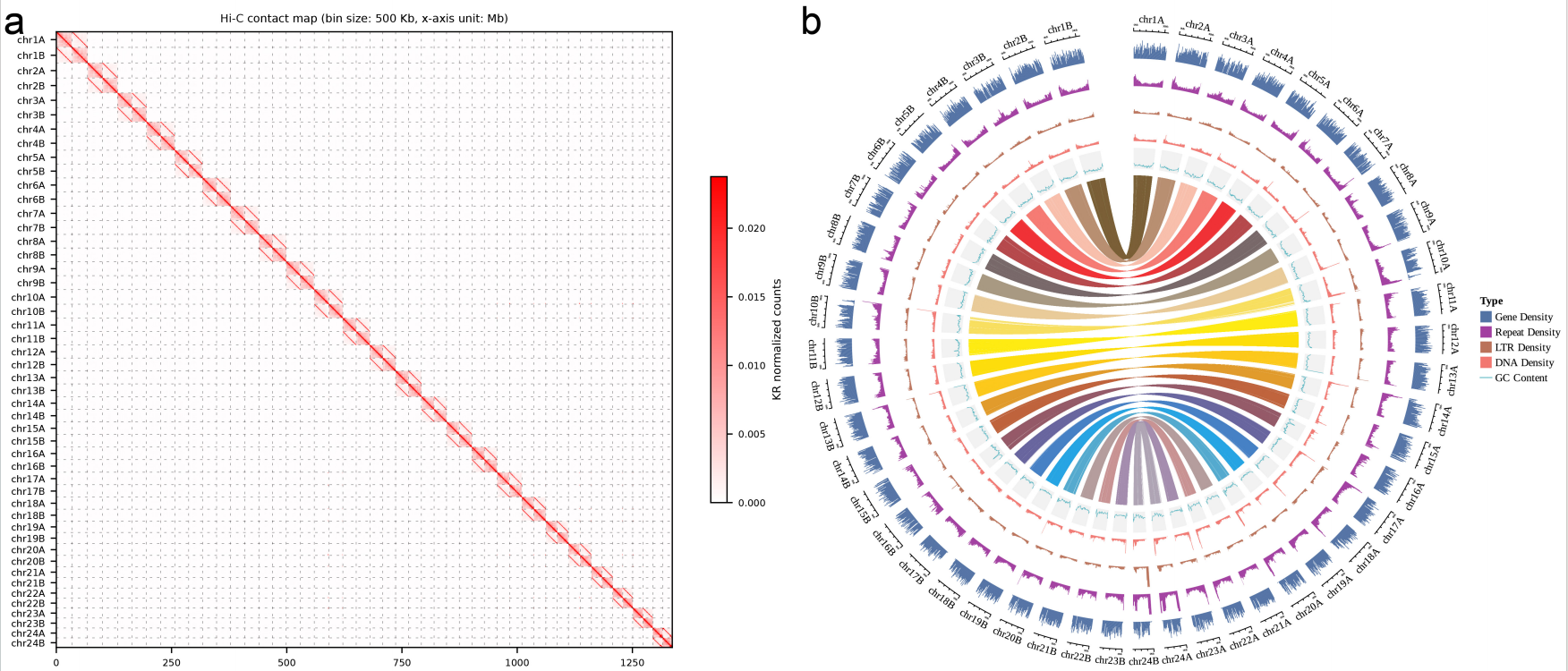
图1:阿纳鲳鲹的两种单倍型解析基因组组装
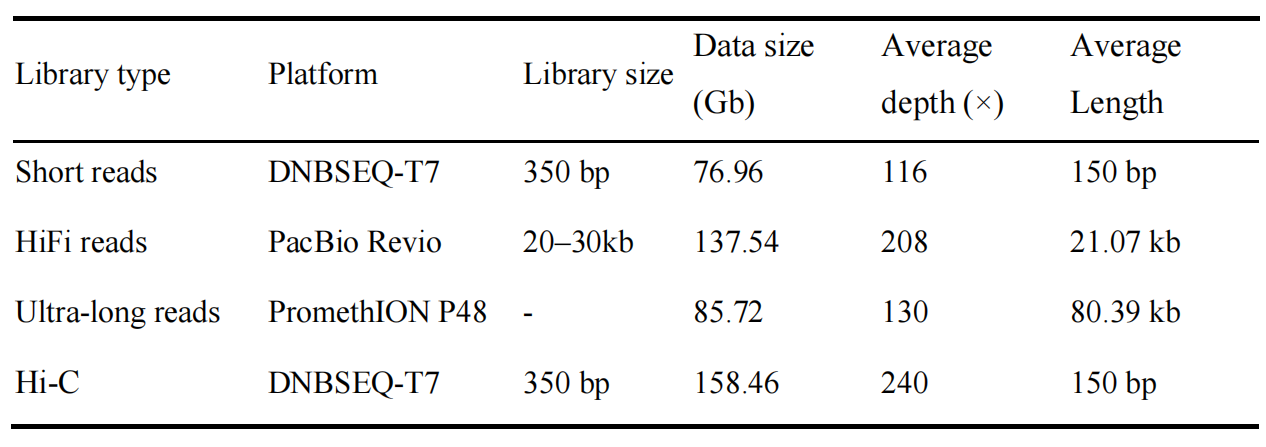
表1:为阿纳鲳鲹基因组组装生成的测序数据
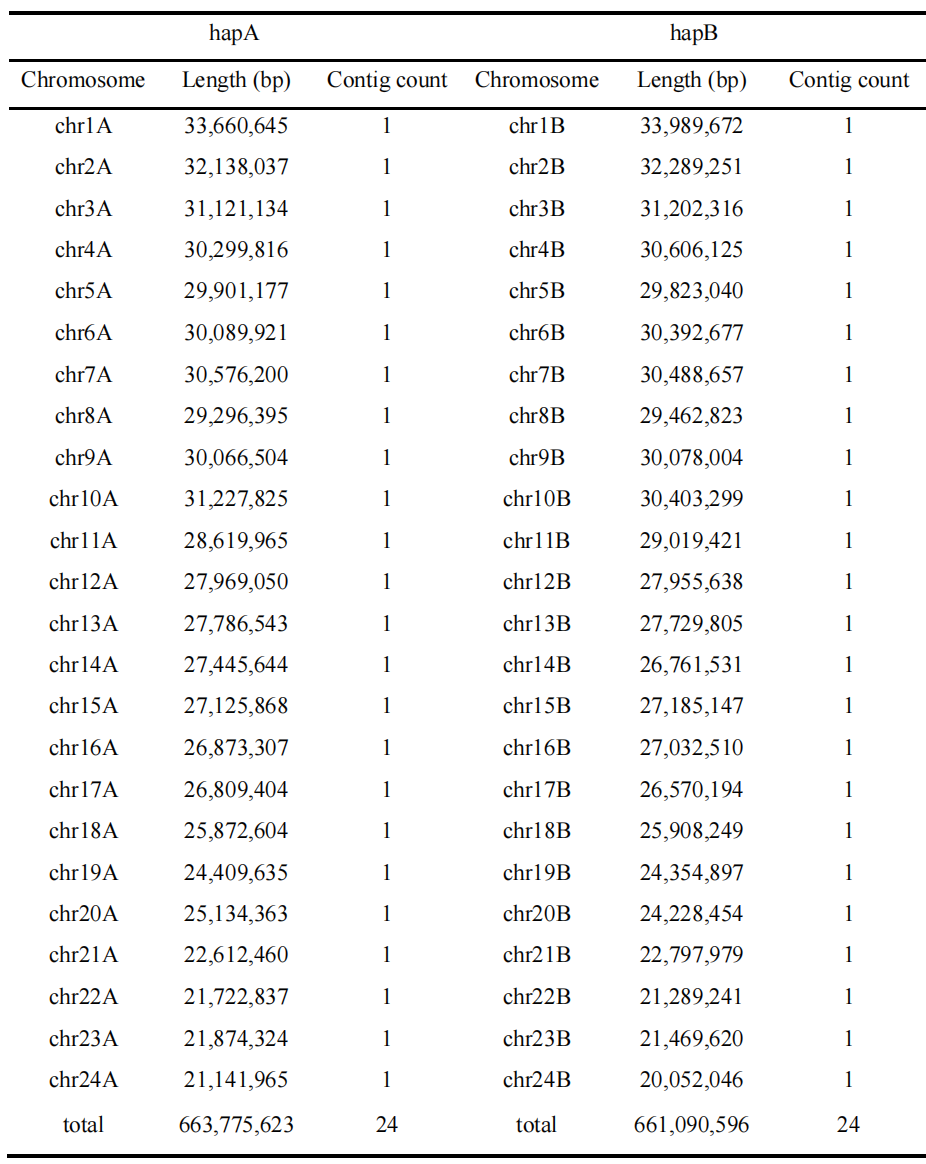
表2:两个单倍型解析基因组的组装统计
二、端粒与着丝粒区域分析
在两套单倍型分辨的基因组组装中,所有染色体两端均成功检测到典型端粒重复序列。在全部 24 条染色体的两端均识别到超过 100 个串联重复的保守 6 bp 端粒基序 TTAGGG,共计 48 个端粒。所有染色体末端均存在规范的端粒结构,表明染色体末端完整,进一步证实了该基因组组装在染色体层面达到了端粒到端粒的无缺口连续性组装。
在着丝粒预测结果中,两套单倍型基因组的 19 条染色体上均成功识别到候选着丝粒区域,而其余5条染色体未检测到明确的着丝粒信号。未被识别的着丝粒区域可能对应于非典型着丝粒结构,其序列组成可能包含复杂的卫星重复家族、长末端重复(LTR)反转座子或其他尚未鉴定的重复元件。
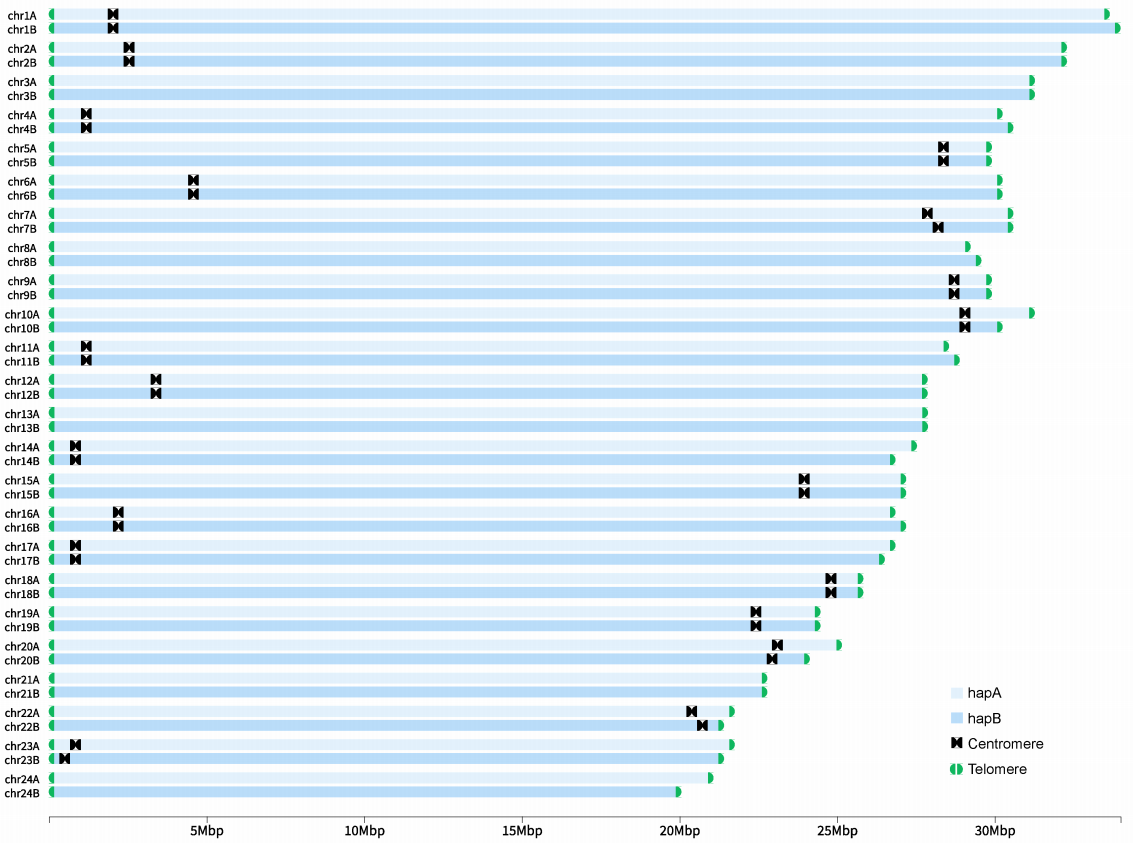
图2:两个单倍型解析组装的端粒和着丝粒展示
三、重复序列与非编码 RNA 注释
在阿纳鲳鲹的两套单倍型基因组中,重复序列占据较大比例。分散重复序列在 hapA 和 hapB 中分别占 24.29% 和 23.09%,串联重复序列分别占 4.02% 和 3.93%。分散重复序列以 DNA 转座子和 LTR 元件为主,DNA 转座子占基因组约 11%,LTR 元件占约 8%,而 LINEs 和 SINEs 所占比例相对较低,表明转座元件在该物种基因组结构中具有重要作用。非编码 RNA 注释结果显示,两套单倍型基因组中均鉴定到大量 miRNAs、tRNAs、rRNAs 和 snRNAs,反映出阿纳鲳鲹基因组在转录调控及细胞基本生命活动中具有较为复杂的调控潜力。
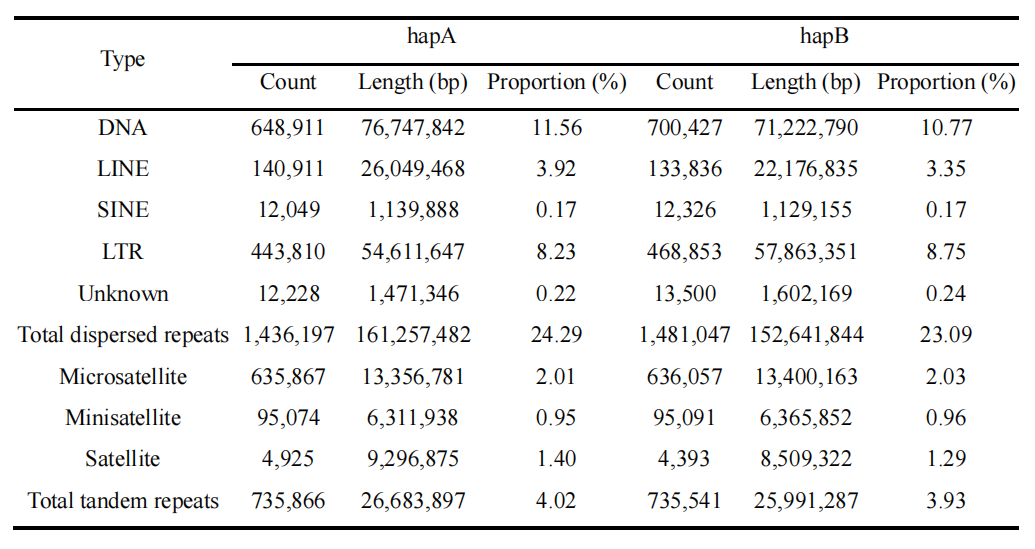
表3:重复序列统计
四、基因预测与功能注释结果概述
在两套单倍型基因组组装中共鉴定到数量一致且结构完整的蛋白编码基因集合。hapA 和 hapB 组装中分别预测得到 23,118个和 23,119个蛋白编码基因,显示出两套单倍型在基因内容上的高度一致性。功能注释结果表明,绝大多数预测基因均可获得可靠的功能信息,其中 hapA 和 hapB 中分别有23069个和23068个基因至少被一个功能数据库成功注释。
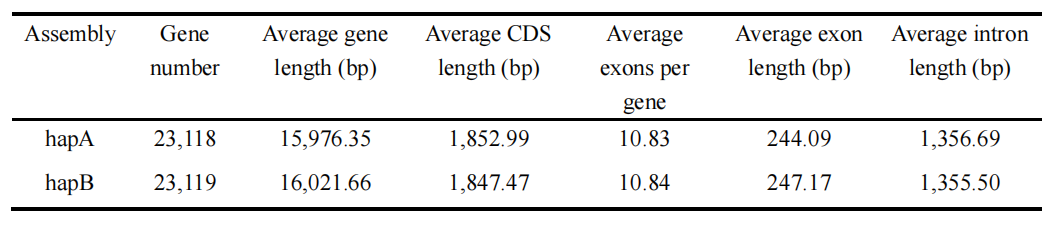
表4:预测蛋白质编码基因统计
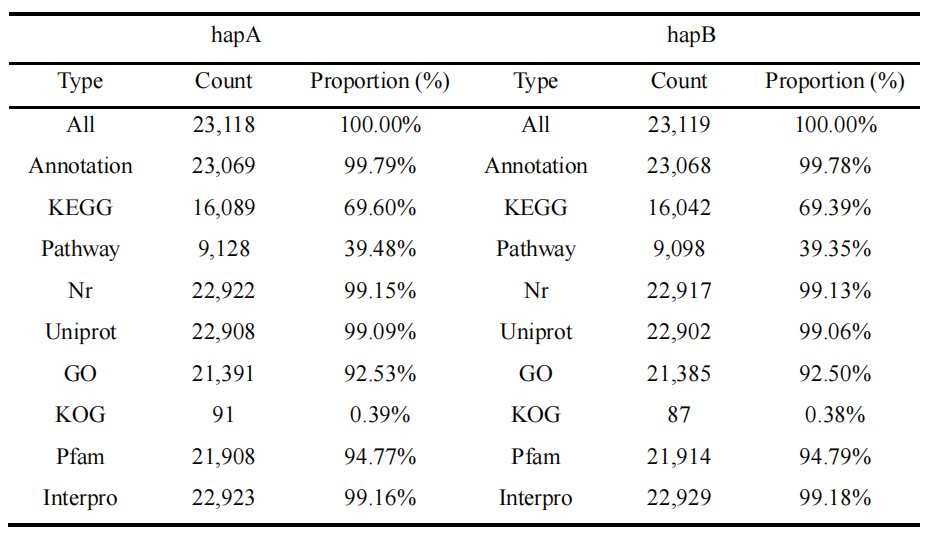
表5:功能注释统计
结语
本研究以阿纳鲳鲹为研究对象,构建了雌性个体的两套单倍型、端粒到端粒无缺口染色体级参考基因组。两套组装分别锚定至 24 条染色体,组装规模约为 661-664 Mb,染色体末端均具有完整端粒结构。组装结果在连续性、完整性和准确性方面均表现优异,质量评估显示基因组具有较高的一致性和完整度。基于高质量组装,对重复序列、非编码 RNA 及蛋白编码基因进行了系统注释,共预测获得约 2.3 万个蛋白编码基因,其中绝大多数获得功能注释。该研究为阿纳鲳鲹的基因功能研究、比较基因组学分析及分子育种应用提供了可靠的基因组基础资源。

